Cell | 深度突变学习预测SARS-CoV-2受体结合域组合突变对ACE2结合和抗体逃逸的影响...
本文介绍一篇来自于苏黎世联邦理工学院的Joseph M. Taft在Cell上发表的工作——《Deep Mutational Learning Predicts ACE2 Binding and Antibody Escape to Combinatorial Mutations in the SARS-CoV-2 Receptor Binding Domain》。
SARS-CoV-2的持续变异以及对疫苗和中和抗体产生耐药性变种的出现,有可能延长COVID-19流行的时间。SARS-CoV-2变种的产生部分是由于病毒突刺蛋白,特别是ACE2,与受体结合域(RBD)的突变产生的。ACE2也是中和抗体的主要靶点。作者提出了一种基于机器学习的蛋白质工程技术——深度突变学习(DML)。DML能对ACE2结合和抗体逃逸的影响做出准确预测,并且能查询数十亿RBD变体组合突变的大规模序列空间。高度多样化的SARS-CoV-2变种已经被确定来自多种进化路线。DML可用于对当前和未来变异的预测分析,包括高突变的变异,如奥密克戎(Omicron),从而指导COVID-19治疗性抗体疗法和疫苗的开发。

1.介绍
在2021年和2022年期间,具有较高传播率和免疫逃逸(抗体逃逸)的SARS-CoV-2变种已经取代了最初的原始株(Wu-hu-1)。这类变体通常在RBD中至少存在一个突变,会直接影响与ACE2的结合。例如,Alpha (B.1.1.7)、Beta(B.1.351)和Gamma (P.1)变异均具有N501Y突变,该突变与ACE2具有较高的亲和力。
中和抗体,包括单克隆抗体疗法和那些由疫苗诱导的抗体(原始的Wu-Hu-1刺突蛋白),通常与变体的结合活性和中和活性会降低。详细的分子分析表明,许多SARS-CoV-2中和抗体具有相同的序列和结构特征,这导致它们被分为四个由RBD靶向表位组定义的常见类别。第1类抗体包括以前临床使用的REGN10933 (casirivimab)和LY-CoV16 (etesevimab)。K417位点突变的循环变异体(例如Beta、Gamma和Delta + (B.1.617.2 + K417N))以及水貂Y453F突变(Cluster 5)会降低这些1类抗体的中和作用。2类中和抗体包括临床使用的LY-CoV555 (bamlanivimab)也会强烈抑制ACE2的结合。然而,具有RBD突变E484K/Q的Beta、Gamma、eta (B.1.525)、Kappa (B.1617.1)和iota (B1.526)等变体都会导致大大降低结合活性和中和活性。第3类抗体,包括临床使用的REGN10987 (imdevimab)和S309 (sotrovimab),结合部分保守表位,并对几种变体(如Alpha、Beta、Gamma)产生耐药性。CR3022等第4类抗体针对sarbecovirus病毒中高度保守的表位,并且在很大程度上对逃逸变体具有抗性,但由于它们不直接抑制ACE2结合,因此通常缺乏中和作用。
Omicron的出现表明,变异可以进化出严重的免疫逃避特性,例如逃脱结合到一系列不同RBD表位的几种中和抗体。值得注意的是,几乎所有临床批准的抗体疗法都失去了对Omicron的实质性中和活性(ba1 / B.1.1.529),包括Eli Lilly(LY-CoV16+LyCoV555)、Regeneron(REGN10933+REGN10987)和AstraZeneca (AZD8895+AZD1061)的多类抗体鸡尾酒疗法。所有这些后来都被美国FDA撤销了临床授权(Eli Lilly和Regeneron)或修改了剂量(Regeneron)。一个例外是S309,它最初是从感染SARS-CoV-1的患者的B细胞中分离出来的,对SARS-CoV-2具有交叉反应性。S309降低了对Omicron的中和活性(ba1),但仍然有效的原因可能是它与在基因多变的sarbecovirus病毒中发现的一个高度保守的表位结合。然而,Omicron亚谱系ba2显示S309大量逃逸,并在2022年初广泛传播,从而导致S309失去临床授权。截至2022年8月,LY-CoV1404 (bebtelovimab)是唯一经临床批准的抗体,该抗体与原始的Wu-Hu-1 RBD结合,对Omicron ba1和ba2变体和新兴的ba4和ba5亚系都保持了很强的中和活性。
Bloom等人对SARS-CoV-2的整个201个氨基酸RBD进行了酵母表面展示和深度突变扫描(DMS),以确定单位置替换对与ACE2结合和逃离单克隆抗体或血清抗体的影响。虽然DMS在RBD单突变分析方面非常有效,但之前的几个循环变体(如Beta、Gamma和Delta)具有多个RBD突变,而Omicron及其亚系具有多达21个RBD突变(BA.1.12.1),因此迫切需要确定组合突变的影响。然而,随着突变数量和氨基酸多样性的增加,组合序列空间呈指数增长,迅速超过了实验筛选技术的能力。例如,当只关注直接参与ACE2结合的20个RBD残基中的一个子集时,理论序列空间远远超过了酵母文库所能筛选出的。
本研究中,作者通过集成RBD突变文库的酵母展示筛选与深度测序和机器学习(图1),建立了深度突变学习(DML)。使用DML全面查询RBD组合突变及其对一组中和抗体中ACE2结合和逃逸的影响,包括临床使用的治疗方法和其他广泛中和和有效的抗体。DML揭示了RBD突变高度多样化的突变范围,这些突变可以保持与ACE2的结合,同时避开许多不同类别的中和抗体。最后,DML能够预测抗体对未来SARSCoV-2变异的稳健性,可以用来评估和选择最有临床发展前途的抗体治疗。
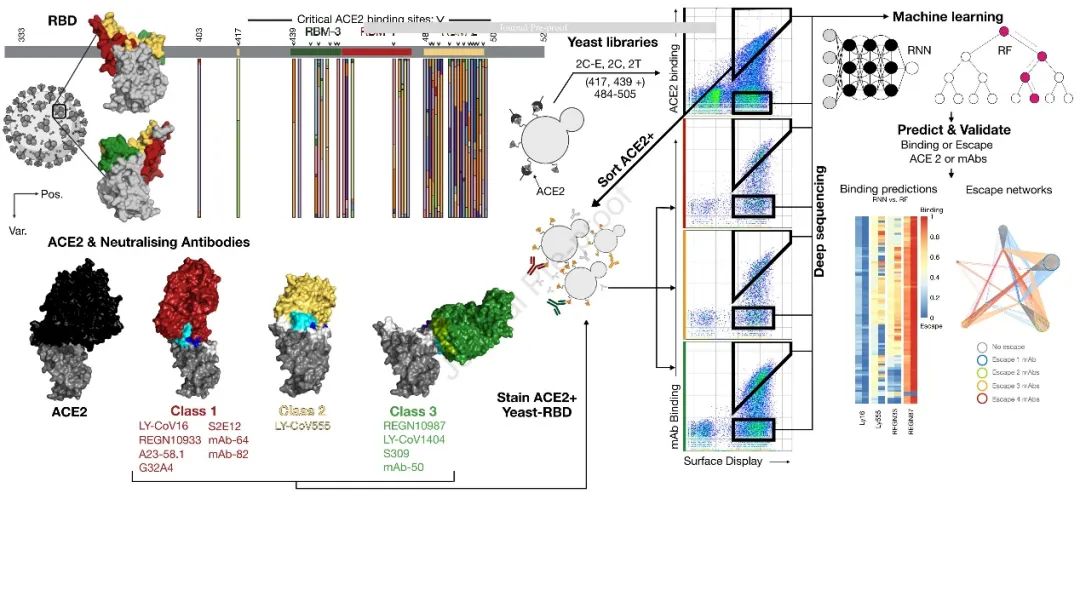
图1 RBD深度突变学习预测ACE2结合和抗体逃逸综述。RBD或SARS-CoV-2刺突蛋白在酵母表面表达,突变文库设计在RBD的受体结合基元上(RBM-3、RBM-1、RBM-2),这些位点与ACE2和中和抗体(如治疗性抗体药物)相互作用。通过FACS筛选与ACE2结合和中和抗体的RBD文库,分离出结合和非结合(逃逸)群体并进行深度测序。训练机器学习模型,根据RBD序列预测ACE2或抗体的结合状态。然后,使用机器学习模型预测ACE2结合和抗体逃逸在当前和未来的变异和谱系。
2.结果
2.1 RBD突变文库的设计与筛选
SARS-CoV-2 RBD突变文库位于受体结合基序核心区域(RBM-3: 439-452位; RBM1: 453 - 478; RBM-2: 484-505)。这是与ACE2接口的RBD子区域,在此区域中病毒基因组测序数据中通常可以观察到突变。为了生成高突变序列空间的训练数据集,Starr等人此前发表了ACE2结合的DMS数据,并设计了组合突变方案。对单个突变的适应度值进行了经验阈值设置并将其转化为氨基酸频率,排除了低于ACE2结合适应度阈值的突变。对于每个位点,通过最小化均方误差选择接近所需氨基酸分布的退化密码子(一些位点由于无法耐受突变和保留ACE2结合而保持固定),从而得到理论氨基酸多样性为 (文库3C)、 (文库1C)和 (文库2C)的RBM文库(图2A)。研究人员还设计了一个扩展版的2C文库,在417位和439位具有完全退化密码子(NNK),这些密码子在许多循环变异体中发生突变,并与抗体逃逸相关,得到的氨基酸理论多样性为(文库2CE)。为了生成较低突变序列空间的训练数据集,作者构建了平铺突变文库,将完全退化密码子(NNK)平铺在每个RBM的3个位置,理论氨基酸多样性分别为 (文库3T)、 (文库1T)和 6(文库2T)(图2B)。
合成的寡核苷酸通过编码不同文库,跨越感兴趣的区域,使用PCR扩增得到与RBD全序列同源的双链DNA。将酵母(S. cerevisiae EBY100)与文库编码的DNA和线性化质粒共同转化,每个文库获得个以上的转化子。RBD变体在酵母表面显示为与Aga2的C端融合。它是基于结合可溶性人ACE2受体,通过荧光激活细胞分选(FACS)分离得到的(图2C)。同时也会分离出与ACE2完全失去结合的RBD变体。重要的是,这并不包括仅部分降低结合的变体,因为这样的中间种群由于不能指定结合或者不结合,不能用来训练监督机器学习模型(图2C)。对所有已排序文库进行RBD基因的深度靶向测序(Illumina),蛋白序列标签显示,ACE2结合和非结合片段的氨基酸使用模式高度相似(图2D)。
抗体包括临床使用的治疗性抗体(REGN10987、REGN10933、LY-CoV16、LY-CoV555、S309和LY-CoV1404),特殊的sabrecoay病毒的抗体(S2E12、S2H97和A23-58.1)和其他直接从COVID-19个体中分离的强效中和剂(G32A4、mAb-50、mAb-64和mAb-82)。每个抗体和文库的结合和逃逸(非结合)的比例变化很大,例如RBM-2文库REGN10933的逃逸突变比例很低,而LY-CoV555的逃逸突变比例很高(图2E)。对所有已排序的RBD文库的抗体结合部分和逃逸部分再次进行深度测序,与ACE2相似,两个部分的蛋白序列标识看起来高度相似。
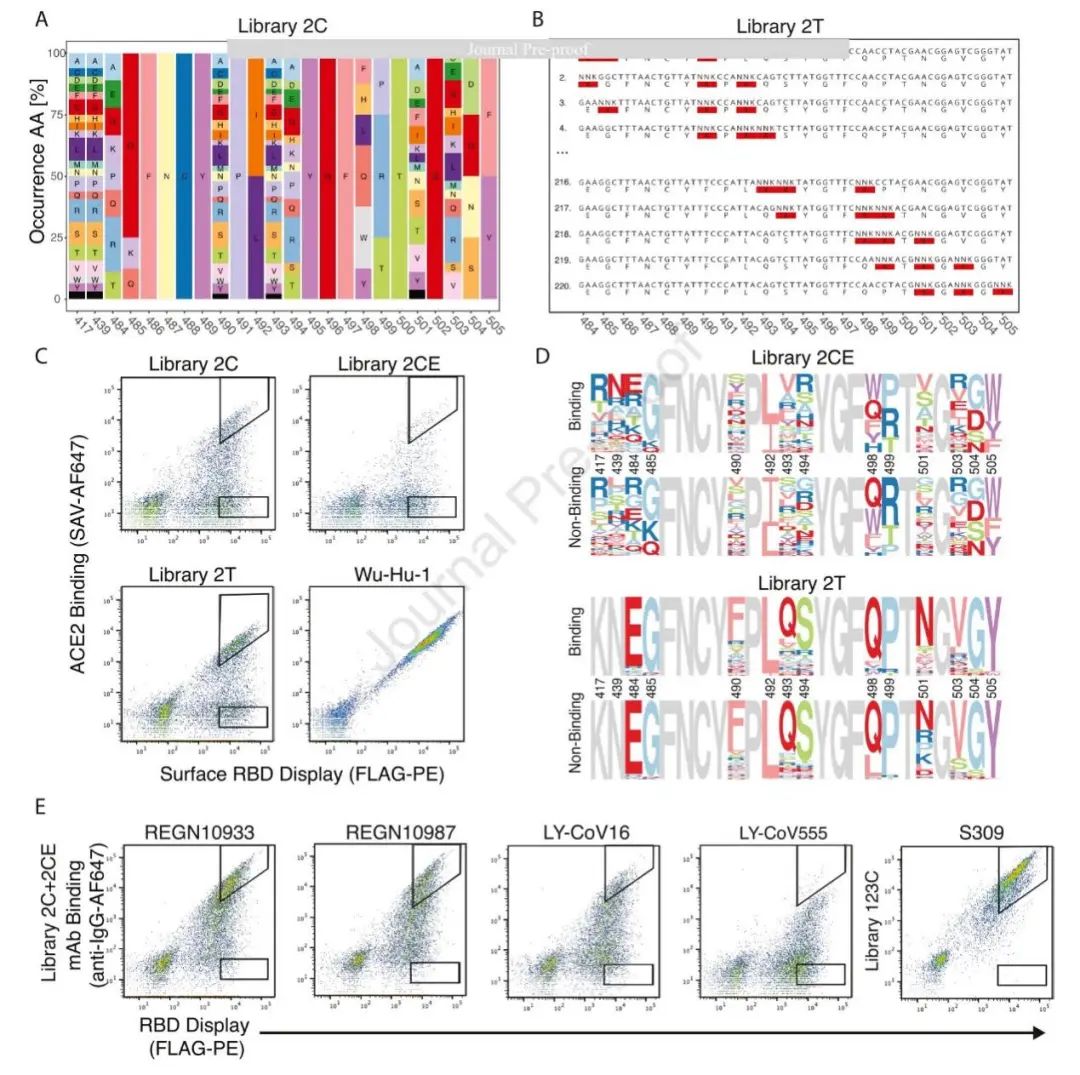
图2 RBD突变文库设计,酵母表面展示筛选及深度测序。(A)显示的是氨基酸在组合文库(文库3C, 1C, 2C)中的使用情况。退化密码子来自于用于ACE2结合的DMS数据 (B) 退化密码子的代表性例子平铺在RBM-2上,它们被汇集在一起组成库2T。(C)流式细胞仪点图显示酵母显示筛选组合(1C、2C、2CE、3C)和平铺RBD文库(1T、2T、3T)和对照RBD (Wu-Hu-1)。(D) RBD的氨基酸标签图是基于ACE2结合和非结合选择的深度测序数据。(E)流式细胞仪点图描绘了酵母展示筛选ACE2结合的RBD库(2C和2CE)的结果。
2.2 机器学习模型能够准确预测ACE2的结合和抗体逃逸
来自ACE2选择的深度测序数据经过预处理、质量过滤和平衡步骤,形成最终的训练集。在核苷酸翻译到蛋白质后,氨基酸序列通过独热编码转换为输入矩阵(图3A)。使用机器学习模型对ACE2结合进行分类,模型输出为任何给定RBD序列与ACE2结合的概率P(较高的P与结合相关)。使用来自RBM-2库的数据训练了一系列基线模型,并使用准确率、F1、精度、召回率进行评估。测试的机器学习模型包括最近邻KNN、逻辑回归、朴素贝叶斯、支持向量机SVM和随机森林RF、RNN。在RBM-2库上训练的所有基线模型都有很好的性能(即准确率在0.87 - 0.94之间)。由于RF和RNN模型具有较好的性能,训练速度较快,作者选择它们进行进一步的优化和应用。
SARS-CoV-2通过一系列突变,包括Omicron等变体及其亚系,它们的RBD中产生了多个组合突变。因此,在不同的突变编辑距离(ED)中确定机器学习模型的性能很重要。最初,由单点突变(ED1)组成的DMS数据被用于训练基线模型,所得到的模型在ACE2结合预测方面表现出非常差的性能(即准确率为0.50, AUC为0.56 - 0.65)。这可能是因为单点突变在较远的距离上不具有可加性,因此无法解释组合突变的非线性影响,导致模型预测几乎所有的组合逃逸变体都是ACE2结合剂。将数据分为低突变距离(≤ED5)和高突变距离(≥ED6)。作者发现,当只使用低距离库(2T)训练模型时,它们对高距离数据ACE2结合的预测性能非常差(准确率<0.65,AUC <0.83)(图3B)。然而,仅使用高距离(RBM-2C/CE)或联合低距离和高距离库(RBM-2 Full)训练的模型在所有距离上的表现都得到了极大的提高,低距离和高距离的准确率分别为>0.94和>0.92,两种模型的AUC均为>0.97(图3B)。
与ACE2选择类似,抗体选择的深度测序数据经过预处理、质量过滤、平衡和编码。使用监督机器学习模型(RF和RNN)对抗体逃逸进行分类,将抗体逃逸定义为给定RBD序列逃逸给定抗体的概率(低P值与逃逸相关)。与之前一样,作者表明,与只有低距离(RBM-2T)或只有高距离库(RBM-2C/CE)的模型相比,同时使用低距离和高距离库(RBM-2 Full)的模型能够更好地预测对典型抗体(LY-CoV16)的逃避(图3C)。几乎所有抗体的RBM-2模型都显示出很高的性能指标,只有LY-CoV555表现出较低的F1得分。机器学习训练和基准测试表明,平衡的分类数据(结合和非结合/逃逸中的序列变体数量相似)是训练精确模型所需的。因此,LY-CoV555模型较低的性能可以通过其不平衡的分类数据来解释(几乎所有RBD变体都逃逸了LY-CoV555)(图3D)。对于RBM-1库,大多数抗体在低ED时产生不平衡的分类数据(很少在≤ED5时产生逃逸变异体)。因此,RBM-1模型仅使用高距离数据(≥ED6)进行训练,除了LY-CoV16、mAb-64和mAb-82这类数据不平衡的抗体(图3E)以外,对大多数抗体都会产生好的效果。最后,对于RBM-3库,几乎所有抗体的分类数据都是不平衡的,因此,未来将排除研究RBM-3机器学习模型。
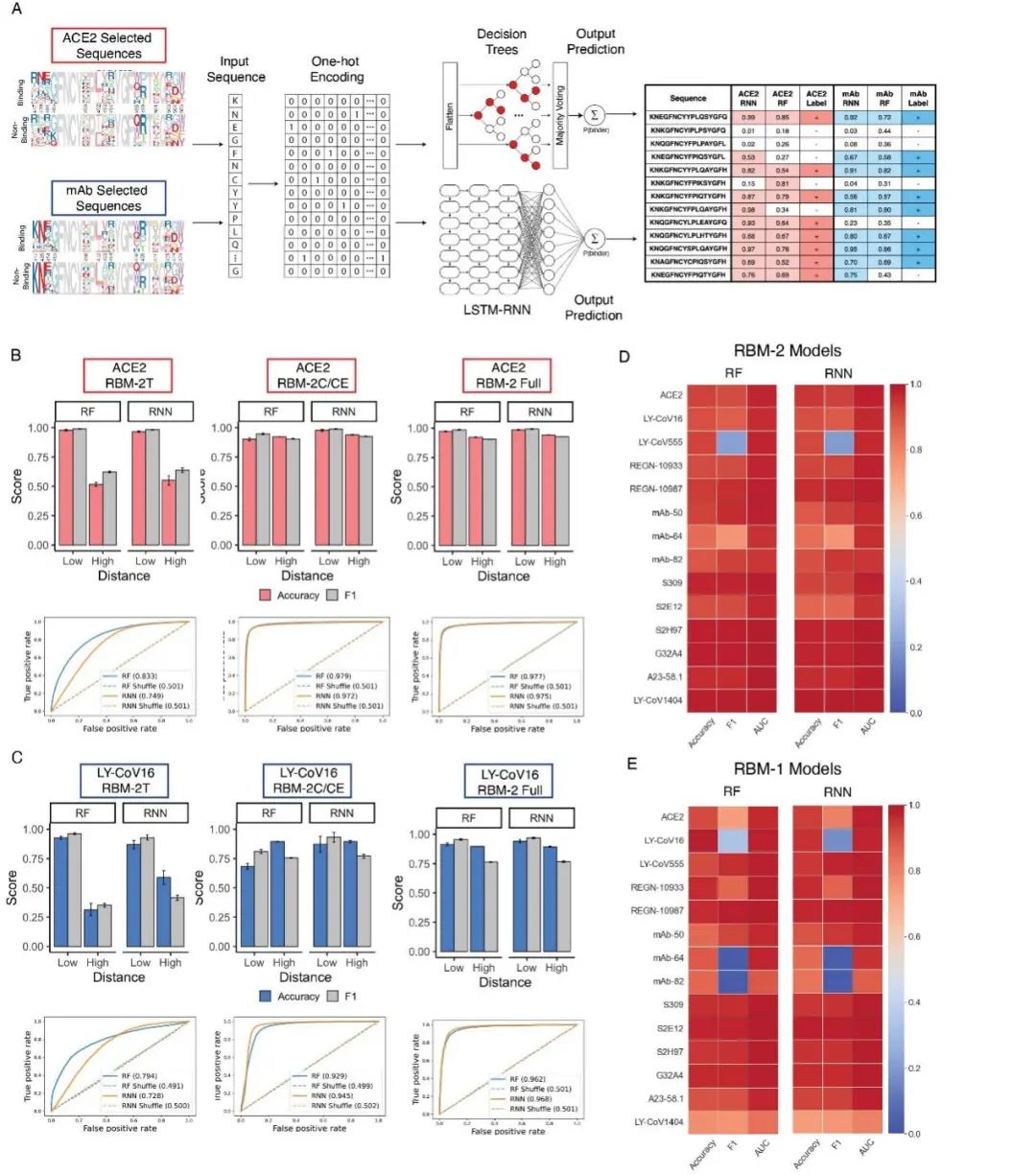
图3 训练和测试基于RBD序列预测ACE2结合和抗体逃逸的机器学习模型和深度学习模型。(A)来自ACE2和单克隆抗体(mAb)选择的深度测序数据经过独热编码,用来训练监督机器学习(RF)和深度学习模型(RNN)。模型根据RBD序列预测ACE2结合或不结合以及mAb结合或逃逸(不结合)的概率(P)来进行分类。(B和C)在2T、2C或Full ACE2或LY-CoV16结合数据上训练的RF和RNN模型的性能,通过精度、F1和ROC曲线显示。低距离序列和高距离序列分别定义为WuHu-1 RBD中≤ED5和≥ED6的序列。(D和E)在RBM-2和RBM-1数据上训练的13个mAb模型的准确性、F1和AUC,在低距离和高距离测试序列上进行评估。
2.3 合成谱系变体的预测分析
用计算机生成合成谱系可以模拟可信的进化路径,其中排除了每个突变步骤中没有预测到ACE2结合中间体的RBD变体(图4a)。重点分析了RBM-2区域和四种中和抗体(LY-CoV16、LY-CoV555、REGN10933、REGN10987)。当RF和RNN模型均得出P > 0.5时,预测给定的RBD序列与ACE2结合,否则预测它们是非结合剂。选择46个合成谱系变体来体现ACE2结合预测的多样性(图5a)。此外,使用类似的模型方法对合成变体的四种治疗性抗体的逃避进行预测。在完成所有的机器学习预测后,将每个合成的RBD变体分别在酵母细胞表面展示,并评估ACE2结合和抗体逃逸情况。模型对ACE2结合的预测准确率为91.67%,非结合预测准确率为100%,总体预测准确率为93.48% (图4b)。对于33个正确预测的ACE2结合变异,4种治疗性抗体的抗体逃逸预测的综合准确率为93.94% (图4c)。AlphaFold2对8个合成RBD变体进行了结构预测(图4d)。结果表明,几个ACE2非结合变体与原始的Wu-Hu-1 RBD没有本质上的差异。相反,ACE2结合变体出现了广泛的结构构象。
图4 RBD变体合成谱系的预测与验证。(A)从Wu-Hu-1 RBD在选定的编辑距离(ED3, ED5和ED7)测试合成变体的工作流程。(B)合成变体的谱系图描绘了基于机器学习 的ACE2结合与非结合的预测和验证 (C)合成变体的点图对应的是机器学习模型(RF和RNN)预测和抗体结合或逃逸的验证。(D) AlphaFold2的结构建模显示了预测的具有ACE2结合(绿色框)或非结合(红色框)的RBD变体结构。
2.4 预测当前和未来变体的抗体逃逸
作者在RBM-2上使用机器学习模型预测ACE2结合谱系上的抗体逃逸 (图5)。REGN10933和REGN10987对Wu-Hu-1、Alpha、Kappa的ED1谱系有很大的适应性(图5A-I)。而Beta和Gamma的ED1谱系几乎完全不受LY-CoV555和LY-CoV16的影响。来自所有变体的ED2谱系中有很大一部分逃逸了REGN10933、LY-CoV555和LY-CoV16,并且随着突变数量的增加,逃逸的可能性越来越大。值得注意的是,小部分的Beta ED2谱系预计会逃脱所有的四种治疗性抗体。其中一些变体在417、484、493和501位点发生突变,这些位点都在Omicron变体中发生突变(图5F)。为了进一步可视化,作者构建了深度逃逸网络(图5C, F, I),描述了四种治疗性抗体对低距离突变的脆弱性。
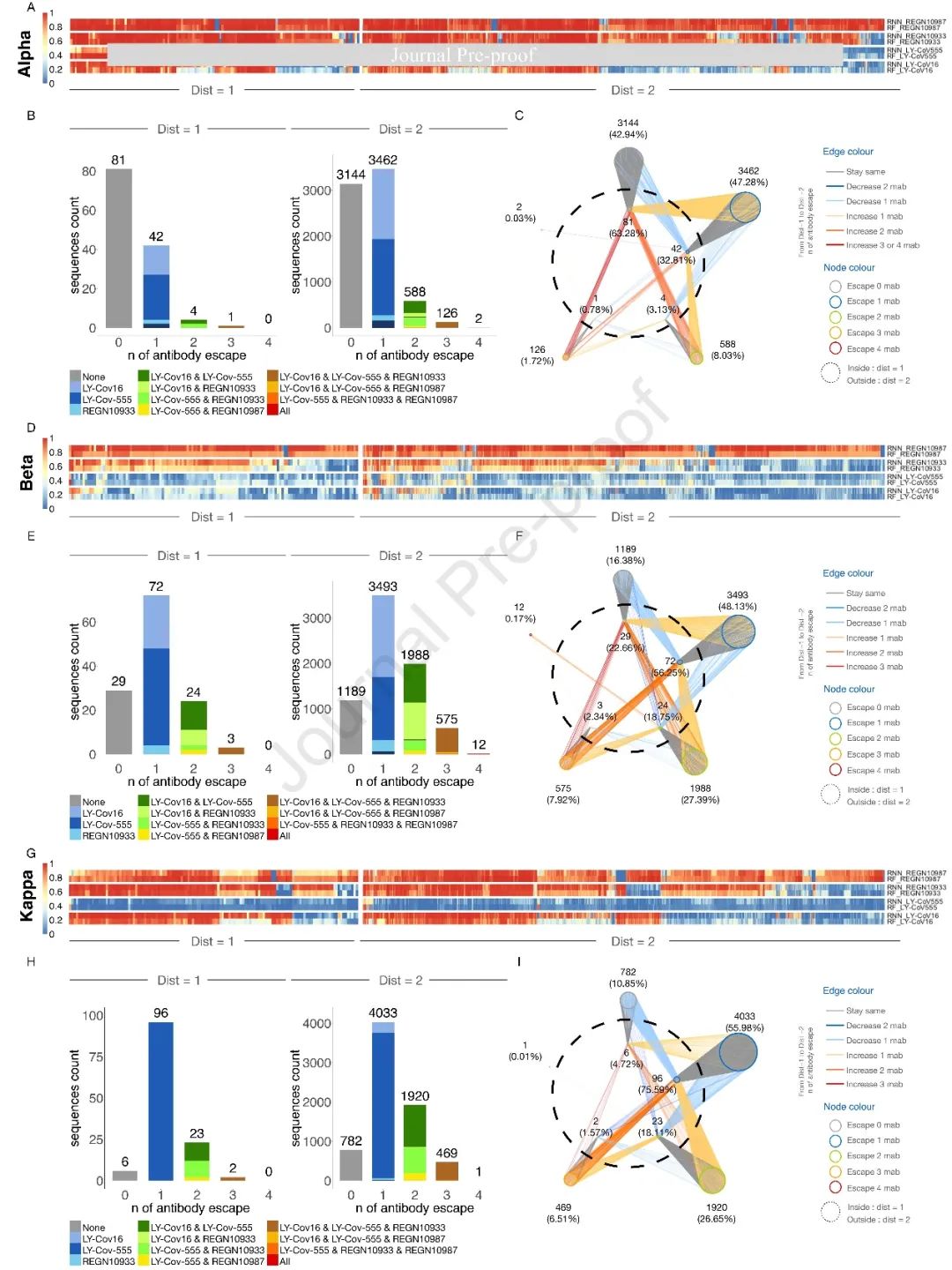
图5 选择RBD变异的低突变距离抗体逃逸的预测分析(A, D和G) 。热力图描绘了单克隆抗体(mAb)结合,通过RF和RNN模型评估Alpha, Beta和Kappa的ED1和ED2变体。(B, E和H)对ED1和ED2转义n个的序列数量(模型间一致,阈值>0.5)。(C, F和I)深度逃逸网络显示了变异及其逃离单克隆抗体之间可能的进化路径。
2.5 确定抗体对SARS-CoV-2突变谱系的稳健性
在选择用于治疗的候选抗体时,抗体对潜在的SARS-CoV-2变异的稳健性(保持结合能力)可能是一个关键参数。为此,作者应用DML来确定是否可以前瞻性地确定几种中和抗体的稳健性。最初,作者专注于与Omicron相对应的合成谱系变体(图6A)。作者测定了RBM-2中Omicron存在的特定单突变和组合突变的抗体逃逸。机器学习显示,一些抗体,如LY-CoV16和LY-CoV555,预计可以保持与大多数单一变异体的结合,但几乎失去与所有组合变异体的结合(图6B),而其他抗体,如REGN10987和LY-CoV1404,预计可以与几乎所有的单一变异体和组合变异体的结合。在此方法的基础上,作者通过计算该位置的突变导致逃逸的平均百分比,探索了给定位置或位置组合中所有突变的影响。这使得作者能够构建动态抗体逃逸谱系,并识别可能导致逃逸增加的突变序列的谱系。作者使用机器学习模型来预测RBM-2中的抗体结合和逃避高距离组合变异体(图6D, E),揭示了几种抗体的不同水平的鲁棒性。

图6 确定抗体对合成RBD变体和突变谱系的鲁棒性。(A)组合文库RBM-2的Omicron (BA.1)突变。(B)在Omicron中观察到的单个和组合突变的结合预测。(C)沿着Omicron谱系的动态逃逸剖面,从Wu-Hu-1距离1-4的所有突变的逃逸序列百分比。(D) Wu-Hu-1编辑距离为6-10中的每个抗体的ACE2结合RBD的抗体预测。
3.讨论
在这项研究中,作者开发了DML,这是一种基于机器学习的蛋白质工程方法,用来确定SARS-CoV-2 RBD组合突变对ACE2结合和抗体逃逸的影响。在DML中,机器学习模型对从文库筛选出数千个标记RBD变体进行训练,可以对数十亿个RBD变体的序列空间进行非常准确的预测,这比仅通过实验筛选的预测要大几个数量级。结合未来的文库设计、基于不同结合阈值的更精密的筛选策略和改进的机器学习模型,可以提高RBD较长长度的预测。作者的DML库是基于原始的Wu-Hu-1 RBD序列,然而,全球几乎所有的循环变体都是Omicron或其亚系。Bloom等人表明不同的RBD变异时,单氨基酸突变(DMS)会导致突变轨迹的改变。考虑到Omicron变体中存在大量的突变,未来的DML研究应使用基于Omicron的RBD序列的突变文库,但需要注意的是,未来可能还会出现其他高距离变体,这需要持续更新文库设计。通过在一个大的突变范围内准确预测抗体逃逸,DML可能使研究人员能够选择最具鲁棒性的候选抗体疗法。未来,除了中和活性,抗体对RBD组合突变的稳健性将是评估COVID-19抗体治疗开发的一个关键参数。最后,有证据表明,其他地方性冠状病毒的受体结合域可能正在经历适应性进化,以逃避人类抗体反应。因此,应用DML来预测抗体逃逸,并结合病毒进化的系统模型,可能有助于识别未来可能出现的变异,从而助力COVID-19疫苗的开发。
4.不足
为了建立DML,作者使用RBD上之前发布的DMS数据,以提高与ACE2结合变体的概率。这对生成足够的机器学习训练数据很重要,但会导致固定一些位置。单突变DMS数据表明,这些位置的突变会导致与ACE2完全失去结合。虽然这种方法在很大程度上有效地覆盖了大多数SARS-CoV-2变体的突变序列空间,但它也存在一些局限性。因为作者的文库设计中的一些固定位置在Omicron或其亚系中发生了突变。最显著的是486位点,该位点在Omicron BA.4和BA.5变体(F486V)中发生突变。此位点与抗体逃逸密切相关。因此,未来的DML突变库设计将需要考虑上位性效应的影响,而不应该只依赖于单突变的DMS数据。此外,在构建库的过程中,作者将RBD分成三个不同的区域来构建。这使得无法探究RBM位点突变的上位性效应。
参考资料
Joseph M. Taft, Cédric R. Weber, Beichen Gao, Roy A. Ehling, Jiami Han, Lester Frei, Sean W. Metcalfe, Max Overath, Alexander Yermanos, William Kelton, Sai T. Reddy, Deep Mutational Learning Predicts ACE2 Binding and Antibody Escape to Combinatorial Mutations in the SARS-CoV-2 Receptor Binding Domain, Cell 2022, ISSN 0092-8674,
https://doi.org/10.1016/j.cell.2022.08.024.
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/126736948
- 点赞
- 收藏
- 关注作者


评论(0)