Hap-eval:Sentieon开源的多测序平台SV精度评估工具
【摘要】 开发背景Sentieon专业软件开发团队除了专注于高效基因组数据分析工具的研发,还与专业合作伙伴一起积极的参与到大型的临床科研项目中,其中就包括了美国国立卫生研究院NIH赞助的Gabriella Miller Kids First (GMKF) 项目。此项目落地于费城儿童医院数据中心,已经产生了来自于2万名患者的4.8万套全基因组数据,涉及了包括44种儿童癌症以及出生缺陷在内的多种疾病。除了...
开发背景
Sentieon专业软件开发团队除了专注于高效基因组数据分析工具的研发,还与专业合作伙伴一起积极的参与到大型的临床科研项目中,其中就包括了美国国立卫生研究院NIH赞助的Gabriella Miller Kids First (GMKF) 项目。此项目落地于费城儿童医院数据中心,已经产生了来自于2万名患者的4.8万套全基因组数据,涉及了包括44种儿童癌症以及出生缺陷在内的多种疾病。除了常规的二代测序之外,GMKF也启动了三代测序项目,评估长读长序列对于基因组结构变异(SV)检测的灵敏度与准确性。相比于二代数据,三代数据不但可以更好的检测到大跨度的结构变异,同时还可以更加准确的提供定相(Phasing)分析。

作为项目的基础,高质量的SV准确率评估工具是必不可少的。然而目前主流的SV评测工具有些主要针对短读长数据,有些无法检测复杂区域尤其是重复区域,而这些需求对于三代长读长SV的准确性评估极为重要。
工具原理
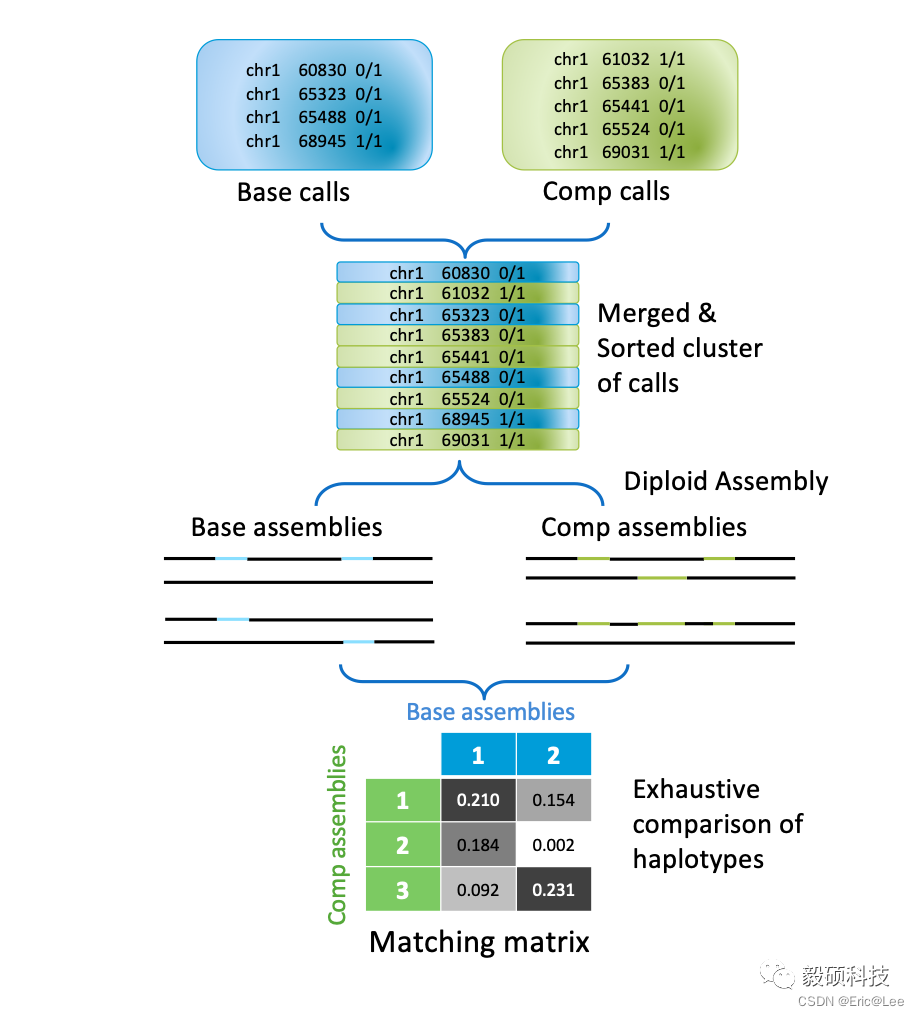
基于项目对效率和精度的要求,Sentieon团队开发了Hap-eval工具,并将之开源。Hap-eval工具是基于单倍型 (haplotype) 对两组SV结果进行比较,首先会将比较区块内的SV拼接成单倍型序列,如果SV的结果中有定相信息,在这一步也可以被利用;然后这些单倍型序列被用来建立一个矩阵,进行结果判断。
安装方法
git clone --recurse-submodules https://github.com/Sentieon/hap-eval.git
pip install ./hap-eval使用方法
usage: hap_eval [-h] -r FASTA -b VCF -c VCF [-i BED] [-t INT] [--base_out VCF]
[--comp_out VCF] [--maxdist INT] [--minsize INT]
[--maxdiff FLOAT] [--metric STR]
optional arguments:
-h, --help show this help message and exit
-r FASTA, --reference FASTA
Reference file
-b VCF, --base VCF Baseline vcf file
-c VCF, --comp VCF Comparison vcf file
-i BED, --interval BED
Evaluation region file
-t INT, --thread_count INT
Number of threads
--base_out VCF Annotated baseline vcf file
--comp_out VCF Annotated comparison vcf file
--maxdist INT Maximum distance to cluster variants (default: 1000)
--minsize INT Minimum size of variants to consider (default: 50)
--maxdiff FLOAT Haplotype difference theshold (default: 0.2)
--metric STR Distance metric (default: Levenshtein)开源地址
https://github.com/Sentieon/hap-eval
【声明】本内容来自华为云开发者社区博主,不代表华为云及华为云开发者社区的观点和立场。转载时必须标注文章的来源(华为云社区)、文章链接、文章作者等基本信息,否则作者和本社区有权追究责任。如果您发现本社区中有涉嫌抄袭的内容,欢迎发送邮件进行举报,并提供相关证据,一经查实,本社区将立刻删除涉嫌侵权内容,举报邮箱:
cloudbbs@huaweicloud.com
- 点赞
- 收藏
- 关注作者

评论(0)