基因组大数据计算: CPU和GPU加速方案深度评测
随着NGS测序通量的大幅提高,搭配高效NGS二级分析技术的精准解决方案快速融进基因组学的各个应用领域:遗传进化、临床诊断、分子育种、医药开发等。以下我们通过对基于CPU和GPU不同硬件平台的NGS二级分析方案进行详细评测,以期为基因组学研究领域的用户提供参考。

Sentieon软件是面向CPU平台开发的,在不需要专用的编程语言,不依赖任何专用硬件的情况下进行快速基因变异检测分析,大幅降低了软件的配置、部署和维护成本。同时,Sentieon针对第三代和第四代Intel Xeon Scalable处理器(以前代号为Sapphire Rapids)进行了改进及优化,更适用于在多核系统上扩展,可以在Xeon平台上实现更快速地处理急诊科及ICU的紧急样本,也可在实验室环境下以更高的通量处理大量样本。

本次评测将比较面向CPU平台的Sentieon软件及面向GPU平台的NVIDIA Clara Parabricks的运算性能和分析准确性,以评估两者在基因组二级分析中的性价比及成本效益。
为什么没选择GATK
GATK是变异检测流程的行业金标准,但它是用Java写的,所以不是性能的金标准。伊利诺伊大学和梅奥诊所发表的文章已经证实,Sentieon 的运算性能明显优于 GATK,而且没有损失精度(https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6710408)。

同时基于最新来自AWS的官方评测,NVIDIA Clara Parabricks相对GATK也有明显的性能优势(https://aws.amazon.com/cn/blogs/hpc/benchmarking-the-nvidia-clara-parabricks-germline-pipeline-on-aws/)
综上原因,我们没有将GATK纳入评测范围。我们的目标是比较Sentieon软件(用C++编写,并针对CPU优化)与 Parabricks(用CUDA编写并针对 NVIDIA GPU 优化)。
测试项目
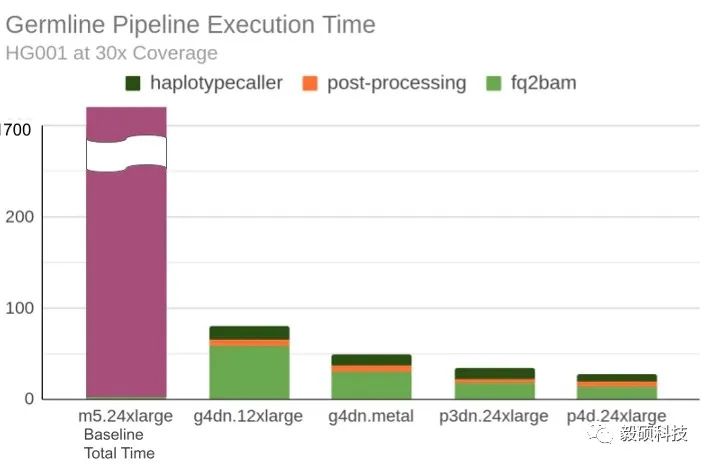
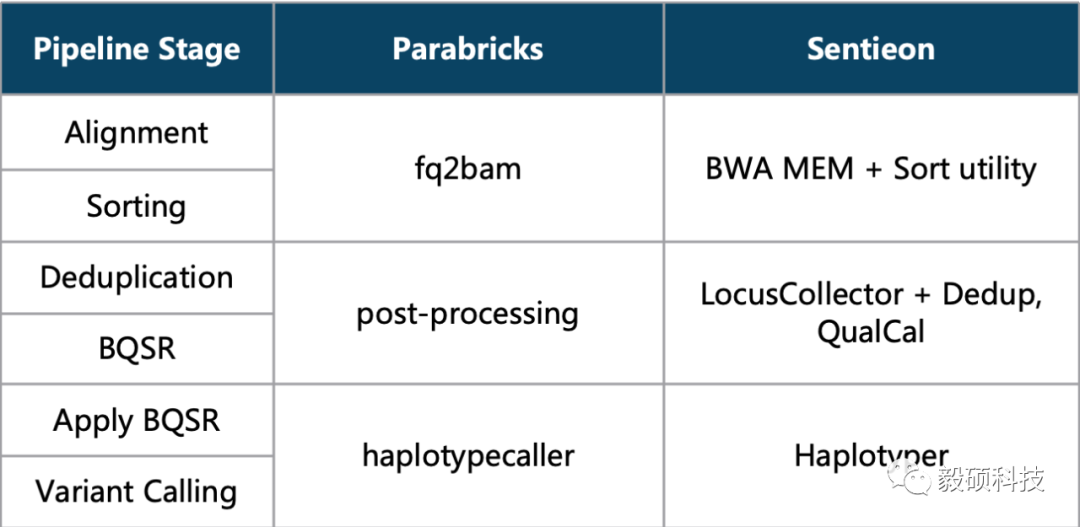
为了尽可能精确地比较Sentieon与Parabricks流程每个计算阶段的运行性能,我们按照两个流程中匹配的计算步骤(如下表),将Parabricks中haplotypecaller、预处理和fq2bam各个阶段对应Sentieon流程的步骤标记出来,并统计两个流程对应计算阶段的运算时间。测试数据为30X HG001 WGS标准数据集。
性能评测
以下我们将展示Sentieon和Parabricks在不同硬件配置环境下的性能表现。其中Intel Xeon 可扩展处理器提供了具有竞争力的性能,第四代 Intel Xeon 可扩展处理器(以前的代号为 Sapphire Rapids)提供最佳的整体性能。除了最重要的性能评测,我们详细对比了每个基因组的计算成本和功耗表现。
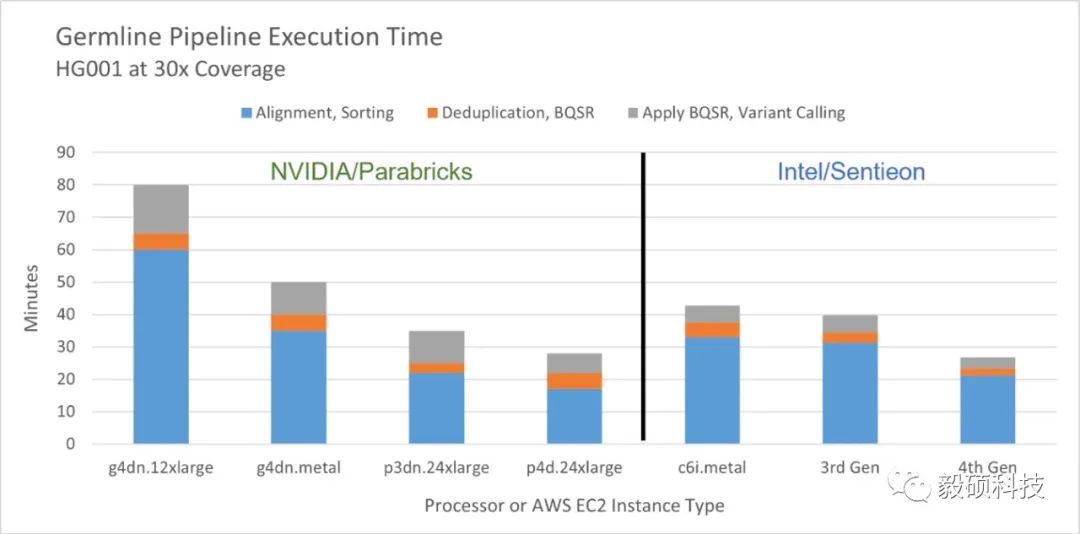
3rd Gen硬件环境为2* Intel 2.4GHz Intel Xeon Platinum 8368 CPU(152核心, 超线程开启),256GB DDR4-3200 内存,1TB Intel 660p 和 2TB DC P4510 SSD。
4th Gen硬件环境为2* 4th Gen Intel Xeon Scalable CPU(原代号为 Sapphire Rapids,>40 核,超线程开启),Intel Pre-production BIOS,256GB DDR内存(16(1DPC)/16 GB/4800 MT/s),1TB Intel D3-S4610 SSD。
3rd Gen和4th Gen的操作系统均为. Ubuntu Linux* 20.04。
通过下表中各计算实例上Sentieon vs. Parabricks的性能对比,可以看出,3rd Intel Xeon平台可在40分钟左右完成30x WGS的数据分析,与GPU平台速度相当。而4th Intel Xeon平台则跑出了26.8分钟的最佳成绩。
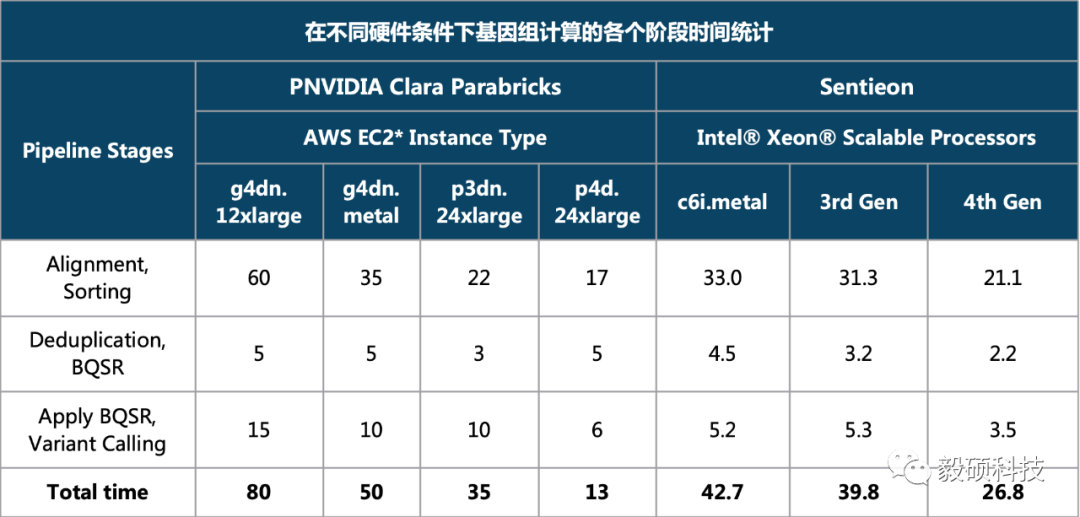
其中NVIDIA Clara Parabricks评测数据来源于AWS官方博客:https://aws.amazon.com/cn/blogs/hpc/benchmarking-the-nvidia-clara-parabricks-germline-pipeline-on-aws/
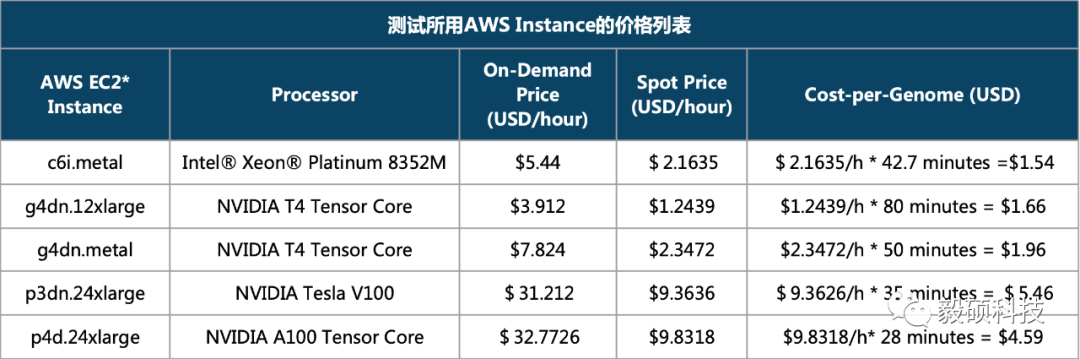
根据不同配置的硬件定价统计表可以发现,与 NVIDIA A100 Tensor Core 处理器(4.59 美元)相比,Intel Xeon Scalable CPU(1.54 美元)的每个基因组分析所需成本要低得多。如果第4代英特尔至强可扩展处理器具有类似的AWS EC2定价,则每个基因组的计算成本将降至不到 1 美元(2.1635 美元/小时 * 26.8 分钟 = 0.97 美元)。在此补充一下,这次基准测试中使用的4th Gen Xeon Scalable CPU是预发布硬件,因此最终产品的性能可能会有所提高。
在功耗方面,c6i.metal 实例中的两颗Intel Xeon Platinum 8352M 处理器的功率是370W,而 p4d.24xlarge 实例中的八个 NVIDIA A100 Tensor Core 的功率则高达3,200W。Parabricks 要达到最佳性能,相较于Intel纯CPU硬件环境需要8.6倍的功率和 3.0 倍的成本,但分析效率相较于Intel 3rd Gen Xeon Platinum 8352M CPU来说,仅有1.5 倍的性能提升。
精度评测
在分析精度方面,Parabricks官方博客此前报道了其变异检测结果的准确性与GATK相当(F1 scores)。而Sentieon不但提供与GATK一致结果(一致性达99%以上),更是PrecisionFDA Truth Challenge的连续冠军。在第二届PrecisionFDA Truth Challenge比赛中更是赢得了4个分项赛冠军。

关于对Sentieon软件的运行效率和准确度的行业认可,其遍布全球的用户和快速增长的数据处理量提供了广泛的例证。

结论
Sentieon软件是通过改进算法模型实现性能加速(纯CPU环境,支持X86/ARM),不依赖于昂贵高功耗的专用硬件配置(GPU/FPGA),不依赖专有编程语言;同时Sentieon软件针对几乎所有的短读长和长读测序平台进行了优化,是FDA多次公开挑战赛的连续赢家。本次评测展现了Sentieon软件在Intel Xeon平台上的卓越性能,是基因组二级分析的最佳解决方案。
- 点赞
- 收藏
- 关注作者


评论(0)