Sentieon实战:NGS肿瘤变异检测流程
肿瘤基因突变检测是NGS的一个重要应用,其分析难点主要在于低频变异的准确性。不同于遗传病检测,肿瘤样本类型多样,测序方法和参数复杂,且缺乏对应各种场景的公共标准真集。再加上常用开源软件经常遇到的准确性低,稳定性差,速度慢等问题。这使得广大诊断公司在评估肿瘤变异检测工具、搭建高效稳定的生信流程上,面临巨大的挑战。
作为NGS数据二级分析的产品专家,Sentieon推出了一系列肿瘤分析流程,适用于从组织样本到液态活检等不同场景。其中,TNscope模块采用Sentieon自主设计的算法,搭配机器学习模型,追求最高的灵敏度、准确度以及使用的灵活性,适合临检样本。TNhaplotyper2模块在结果上匹配GATK最新4.1版本的Mutect2,同时提速10倍以上,更加适用于科研样本。
本期分享近期发表的3篇文献,对TNscope和TNhaplotyper2的性能和准确性进行的详细和客观的评估。最后我们将展示BMS(百时美施贵宝)使用Sentieon TNscope检测MNV纠正TCGA数据库的案例。

TNscope
TNscope模块对于低频变异的检测灵敏度非常高,搭配丰富可调的过滤参数或者机器学习过滤器,可以在临检样本上达到很高的灵敏度和特异性。目前,TNscope流程已被业内多家诊断公司采用,在室间质评等质控场景下均有优异表现。
2020年7月由中山大学和安诺优达联合发表的一篇文章中,作者建立测试数据集,系统的比对了目前流行的包括TNscope在内的多款肿瘤变异检测软件,从准确度和速度两方面给出了评测数据。

文中作者使用细胞系真集合成不同肿瘤纯度的真集数据,来评估不同的肿瘤变异检测流程。在这里,作者将HG001和HG002 两个标准品原始reads按不同稀释比例(10%-60%)混合,合成了4套100x深度的模拟数据,经过比对,去重,重比对,BQSR等步骤后,用6款变异检测软件进行变异检测,包括TNscope (v201808.05),TNseq (v201808.05,匹配3.8版本MuTect2结果),Mutect2 (v4.0.7),NeuSomatic (v0.2.0),VarScan2 (v2.3.9),Strelka2 (v2.8.4)。输出结果经过RTG Tools和真集比对计算Precision和Recall。
在区分度较大的5%变异丰度数据集中,我们可以看到, TNscope和Mutect2 (和TNhaplotyper2结果一致) 在SNP和Indel检测上都大幅度领先其他软件,尤其是TNscope能够在基本杜绝假阳性的基础上达到较高的灵敏度。
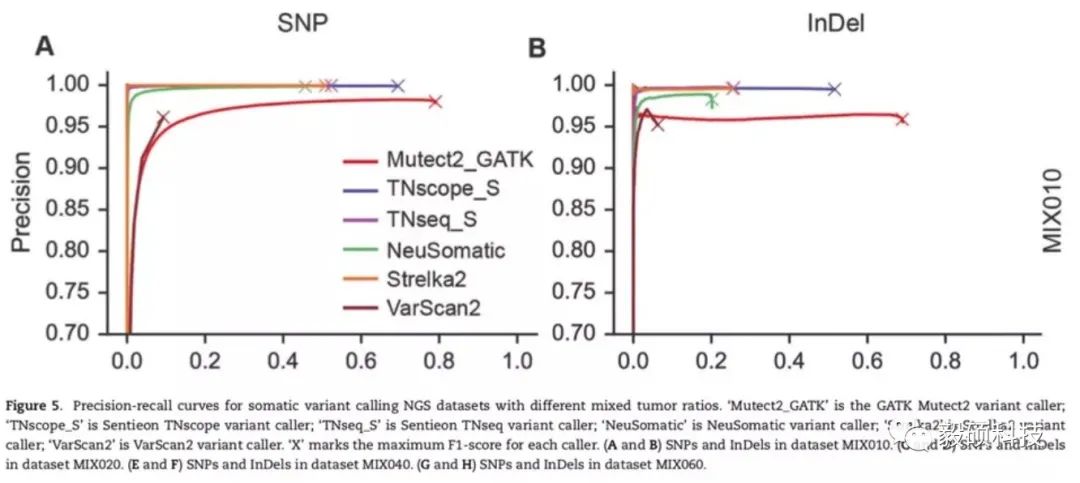
除了准确度以外,生信流程的分析速度是临检环境的另一个关注点,尤其是随着样本量的增长,高深度大panel的普及,单个样本测序数据量也在飞速增长。提高分析速度,加快产品交付周期变得至关重要。本文比较了不同软件的计算时长:TNscope的速度遥遥领先,和Mutect2相比提速10倍左右。
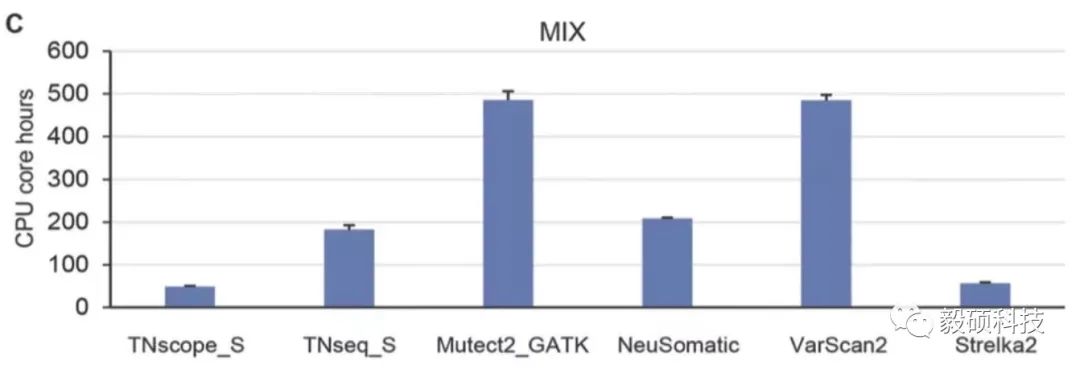
综合本文中准确度和速度两方面的指标,Sentieon的TNscope性能超越了其他所有参评的软件模块。我们期待TNscope能够助力更多的临检用户,在提升分析准确性的同时大幅提升速度,压缩计算成本。
TNhaplotyper2
在科研领域,GATK的Mutect2以其优异的性能和Broad Institute的强力背书一直被业内作为金标准所接受。进入GATK4时代以来,Mutect2进入了技术尝试期,在子版本更新中,频繁更新算法,大量引入新参数,相邻子版本的变异检测结果差异有时会高达50%。这样的大幅变化,给用户带来了很大困惑,对Mutect2的稳定性也产生怀疑。因此,Sentieon在匹配了4.0.2版本之后也暂停了对不稳定Mutect2版本的匹配更新。进入4.1版本之后,Mutect2 完成beta更新测试,在完善功能模块的同时,版本更新也逐步稳定。所以,Sentieon应广大用户的要求,发布了匹配目前最新的4.1.9版本的TNhaplotyper2加速方案。
2020年7月份,在BMC Medical Genomics上发布了的一篇文章,对4.1版本的Mutect2进行了性能测试。作者选取了同一个组织样本的新鲜冷冻(Fresh Frozen)样本以及福尔马林固定切片(FFPE)样本进行了NGS测序,对结果数据进行肿瘤突变检测。随后以新鲜冷冻样本为金标准,考察不同检测软件流程在处理FFPE样本时的准确度表现。

文章选用了四个常用的软件,包括了Mutect2 (v4.1.2),VarScan2 (v2.2.3),Shimmer (v0.2),以及Strelka2 (版本未汇报)。作者首先使用每个软件检测新鲜冷冻样本FF的变异,作为比较的基准,然后检测FFPE样本的变异,并和FF的结果进行比较。比较的方法是通过调整每个软件最有代表性的肿瘤概率annotation,例如Mutect2用TLOD,Strelka2使用EVS等,这样在生成ROC curve上找到F1最大值,作为每个软件的优化结果。
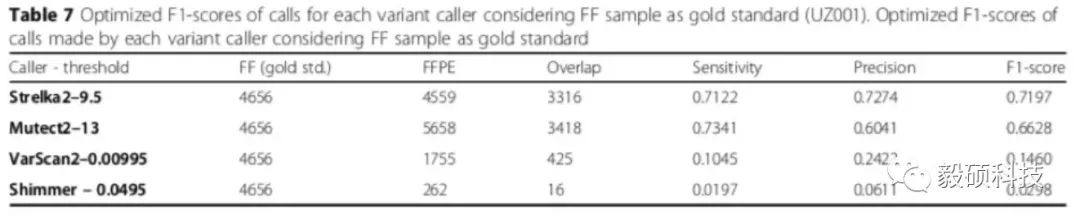
从上图中可以看出,Mutect2和Strelka2给出的FFPE结果比较接近,说明能更好的区分FFPE的artifact,远远优于VarScan2和Shimmer的结果。作者在讨论中也着重指出了,不同检测软件的选取对于突变结果的影响甚至超过了样本类型的影响,这也再次证明了软件的性能对于最终结果准确性的巨大影响。
值得注意的是,文中使用的4.1.2并不是当前的最新版本,Sentieon TNhaplotyper2所匹配的版本4.1.9的准确性会比文中所显示的更加优异。同时,TNhaplotyper2运行时间仅为Mutect2的1/10,大大缩短了项目周期,节约了计算成本,是科研用户的最佳选择。
MNV肿瘤合并
本篇文献来自于药企BMS(百时美施贵宝),BMS与Sentieon团队合作,利用Sentieon的肿瘤MNV合并模块对TCGA等公共数据库里的结果进行检查,发现了大量VCF结果中的MNV被错误标记为了SNV,导致氨基酸翻译错误,继而影响了后续的医学诊断。

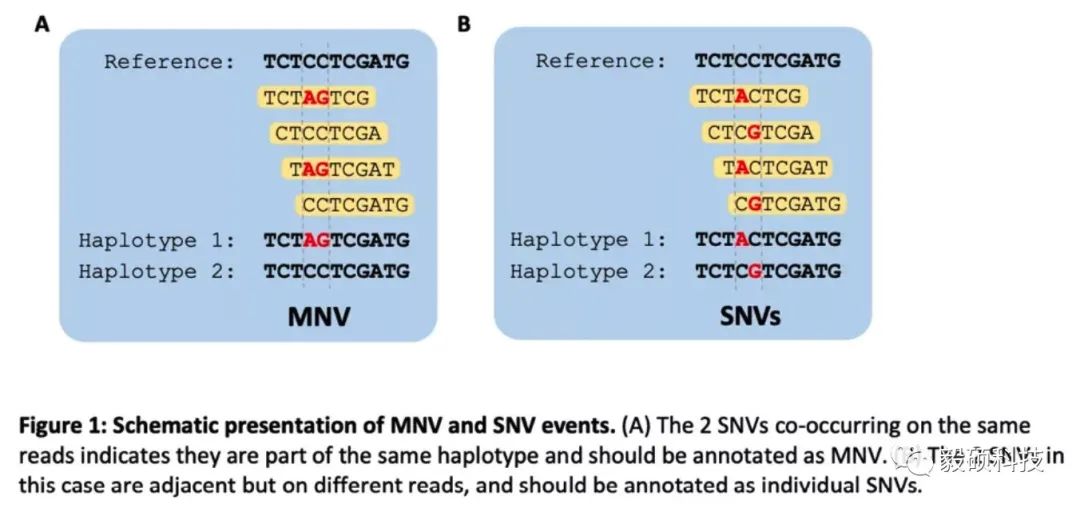
要想准确的鉴定MNV复杂变异,Haplotype的识别是第一步,这就要求变异检测软件必须进行局部组装(local denovo assembly),利用reads的信息拼接出不同的haplotype,从而确定不同位置突变之间的连锁关系。Sentieon的TNscope以及TNhaplotyper2模块都会输出haplotype信息。如果两个相近的变异位于同一个haplotype上,那么Sentieon的MNV合并模块将会把它们在VCF中合并成一个MNV。
在TCGA数据集的重新验证中,文章发现12,141个MNV被错误注释了,另外有7个常见突变基因的MNV在20个数据集中都没有被正确合并。最常见的一个例子就是BRAF V600K变异,在若干公共数据集中都是错误的作为BFAF V600E和BRAF V600M的SNV汇报。类似的例子在TCGA数据集中频繁出现,下图是一个数目的汇总。
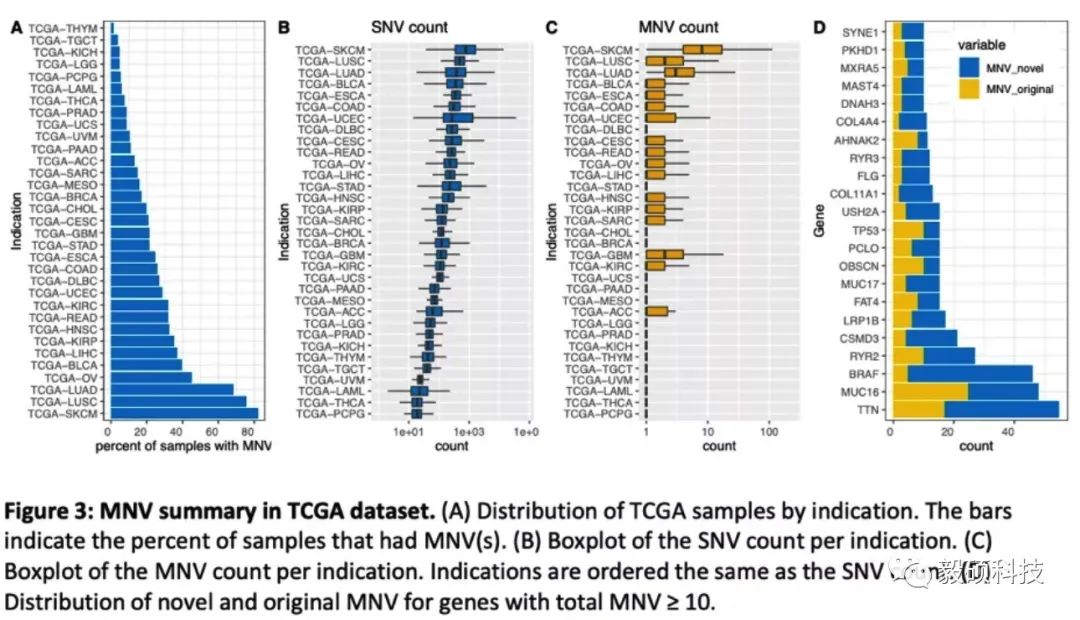
文末总结道,虽然自2017年以后的大型项目中的MNV都已经被正确的合并了,但是很多中小型学术项目或者商业公司的数据集里,MNV合并还是经常被遗漏,有待修复。只有正确判断出MNV才能判断出正确的蛋白氨基酸变异,作者在此推荐在任何肿瘤流程中都使用类似于Sentieon MNV这样的合并工具。
软件试用:https://www.insvast.com/sentieon
- 点赞
- 收藏
- 关注作者


评论(0)