R语言之可视化-火山图
【摘要】 火山图可以方便直观地展示两个样本间基因差异表达的分布情况。通常横坐标用log2(fold change)表示,差异越大的基因分布在两端,纵坐标用-log10(pvalue)表示,T检验显著性P值的负对数。通常差异倍数越大的基因T检验越显著,所以往往关注左上角和右上角的值。我们根据计算结果在图上按照FDR和我们自定义的logFC值来将这部分显著变化的基因用不同颜色标示出来以区分...
火山图可以方便直观地展示两个样本间基因差异表达的分布情况。通常横坐标用log2(fold change)表示,差异越大的基因分布在两端,纵坐标用-log10(pvalue)表示,T检验显著性P值的负对数。通常差异倍数越大的基因T检验越显著,所以往往关注左上角和右上角的值。我们根据计算结果在图上按照FDR和我们自定义的logFC值来将这部分显著变化的基因用不同颜色标示出来以区分,这类图像往往呈现类似火山爆发的样子,于是就被叫做“火山图”(volcano plot)了。
-
library(ggplot2)
-
diff <- read.csv("DEG.Out.csv",header = T)
-
-
logFC <-diff$logFC
-
adj <- diff$FDR
-
-
data <- data.frame(logFC=logFC,padj=padj)
-
data$sig[(data$padj > 0.05|data$padj=="NA")|(data$logFC < 0.5)& data$logFC > -0.5] <- "no"
-
data$sig[data$padj <= 0.05 & data$logFC >= 0.5] <- "up"
-
data$sig[data$padj <= 0.05 & data$logFC <= -0.5] <- "down"
-
-
# 选最大值作为xlim的上下边界
-
x_lim <- max(logFC,-logFC)
-
# 绘制火山图
-
library(ggplot2)
-
library(RColorBrewer)
-
pdf(file = "miRNA_volcano.pdf",width=8,height=8)
-
theme_set(theme_bw())
-
p <- ggplot(data,aes(logFC,-1*log10(padj),
-
color = sig))+geom_point()+
-
xlim(-5,5) + labs(x="log2(FoldChange)",y="-log10(FDR)")
-
p <- p + scale_color_manual(values =c("#0072B5","grey","#BC3C28"))+
-
geom_hline(yintercept=-log10(0.05),linetype=4)+
-
geom_vline(xintercept=c(-0.5,0.5),linetype=4)
-
p <- p +theme(panel.grid =element_blank())+
-
theme(axis.line = element_line(size=0))+ylim(0,15)
-
p <- p +guides(colour = FALSE)
-
p <- p +theme(axis.text=element_text(size=20),axis.title=element_text(size=20))
-
p
-
dev.off()
-
print(p)
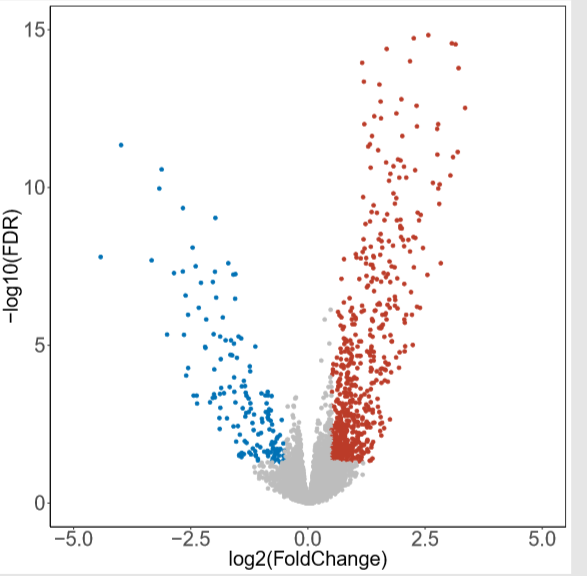
参考:
https://www.jianshu.com/p/efb41e5f0ee2
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/88609851
【版权声明】本文为华为云社区用户转载文章,如果您发现本社区中有涉嫌抄袭的内容,欢迎发送邮件进行举报,并提供相关证据,一经查实,本社区将立刻删除涉嫌侵权内容,举报邮箱:
cloudbbs@huaweicloud.com
- 点赞
- 收藏
- 关注作者

评论(0)