KDD 2022 | 编程指南:生命科学中的图神经网络
本文介绍由亚马逊的研究团队推出的应用于生命科学的图神经网络指南《Graph Neural Networks in Life Sciences: Opportunities and Solutions》,这个工作发表在2022年数据挖掘顶会KDD上。图结构数据在生命科学以及医疗场景无处不在,最近很多研究把原来依赖于描述性数据分析的问题转化成依赖于生物网络的问题,例如图神经网络 (GNNs)。与其它领域相比,生命科学的问题有其自身的独特性和细微的差别。首先,这份指南比较全面地介绍了生命科学中的各种图结构数据,基于这些数据的生物和医学问题,以及相关的基于图机器学习的算法;随后,作者提供了四类基于GNN的解决方案的编程指南,每一种方案都提供了python代码和比较详细的说明,这四类问题包括:1)小分子属性预测;2)大分子属性和功能预测;3)基于双图(bi-graph)的蛋白质-配体对亲和力预测;4)利用知识图谱进行医学预测。所有代码都基于深度学习库DGL-lifesci和DGL-KE。
图机器学习在生命科学的应用
图结构数据在生命科学中十分常见。例如在蛋白质相互作用图中,结点表示蛋白质,边表示蛋白质之间的物理联系;在药物相互关系图中,每个结点是一种药物,边表示药物之间的协同或者对抗作用;在细胞网络中,结点可以表示细胞、肿瘤以及淋巴,边表示他们之间的空间邻近关系;在非编码RNA调节网络中,结点表示非编码RNA,边表示RNA之间的调节相互作用(regulatory interaction);在疾病关系网络中,结点表示疾病,边表示疾病之间的共存性;在医疗保健知识图中,结点是某种医药或者治疗方案,而边代表这些治疗手段的共存关系;把上述的所有图连接在一起,又可以组成一个更大的图中图,在这个大图中每个结点又都是一个图(图1)。基于上述的生物网络图,图机器学习可以用来预测分子的性质、功能,生成具有某类性质的新分子,预测分子之间的影响;预测RNA序列数据疗法的扰动效应,药物再利用,预测药物冲突;利用生物知识图谱进行疾病诊断,辅助临床治疗等等。
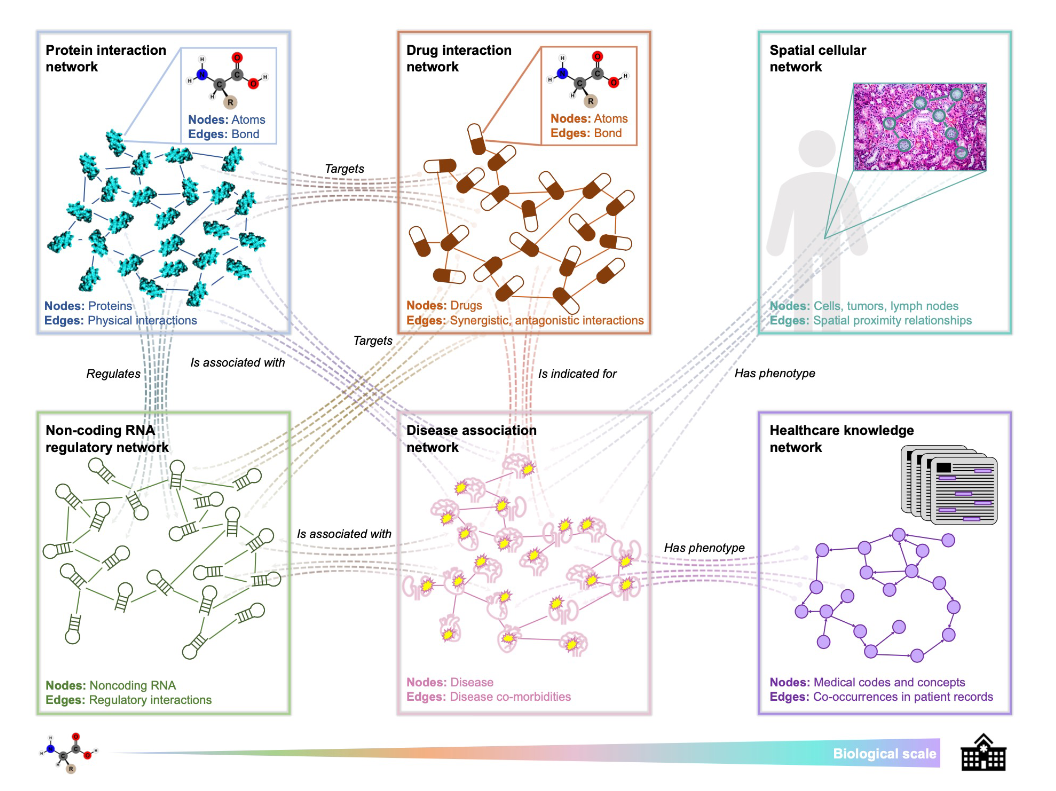 图1
图1
编程指南
作者提供了四类实用编程指南(Github链接在本文末尾)。
 图2
图2
-
利用简化分子线性输入规范(SMILES)构建分子图,从零开始构建GNN模型进行分子属性预测。
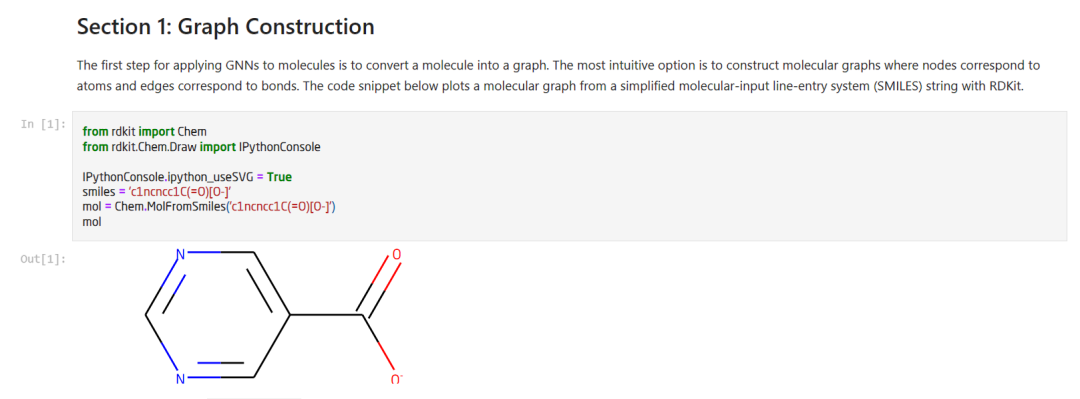 图3
图3
-
在大分子数据RNA和蛋白质上:1)利用GCN预测COVID-19 mRNA疫苗的降解;2)利用等变GNN预测蛋白质功能。作者还提供了实用的RNA和蛋白质结构可视化代码(图4和图5)。
 图4
图4
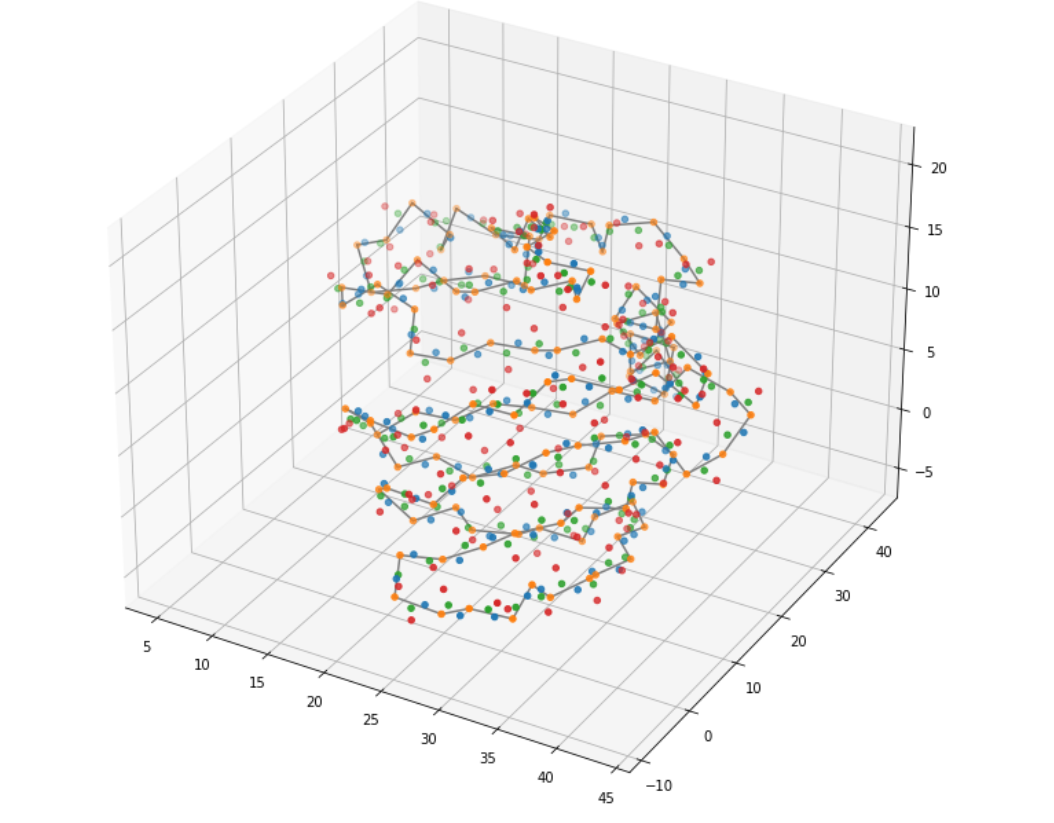 图5
图5
-
基于双图的蛋白质-配体对结合亲和力预测,这个问题研究如何在一对图之间进行预测。蛋白质配体结合亲和力预测对于药物的发现和早期的药物筛选非常重要。作者通过构建PotentialNet进行这项任务。
-
医学知识图谱(KG)的应用。知识图谱表示网络中实体与实体的相互关系,实体可以是对象、事件、情境或抽象概念。作者介绍了医学知识图谱上的三个经典应用:1)挖掘医学文献和出版物中的知识;2)处理来自医学数据库中的不同KG;3)将关系数据库转化为KG。此外,作者介绍了药物再利用知识图谱(DRKG)(图6),DRKG是一个综合性的生物知识谱图,涉及基因、化合物、疾病、生物过程、副作用和症状。
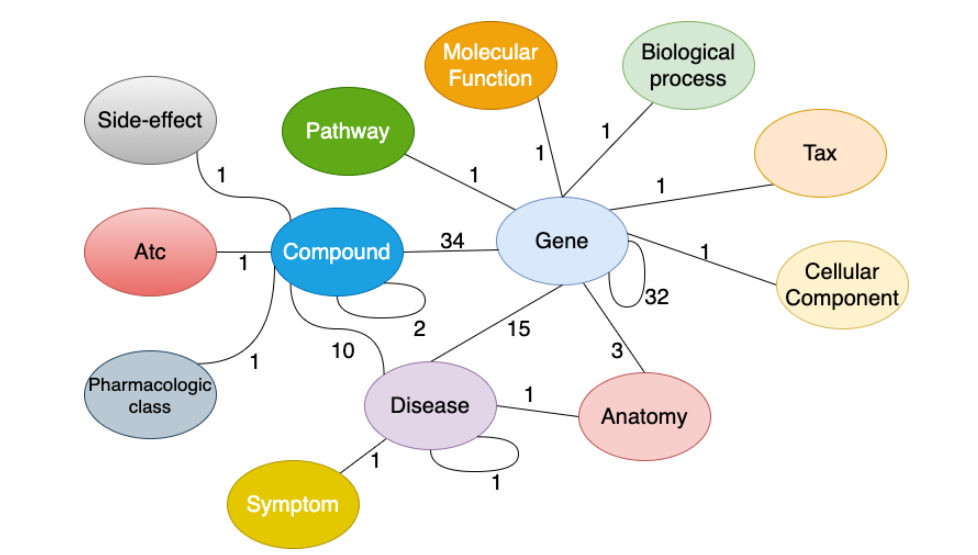 图6
图6
总结
这个工作比较全面地介绍了生命科学中的图结构数据和蕴含在这些数据中的科学问题,并提供了从零开始的编码示例和比较详细的代码说明,为新的相关从业和研究人员提供了很好的入门指导。
参考资料
Wang, Zichen, et al. "Graph Neural Networks in Life Sciences: Opportunities and Solutions." Proceedings of the 28th ACM SIGKDD Conference on Knowledge Discovery and Data Mining. 2022.
代码
https://github.com/dglai/Graph-Neural-Networks-in-Life-Sciences
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/126736968
- 点赞
- 收藏
- 关注作者

评论(0)