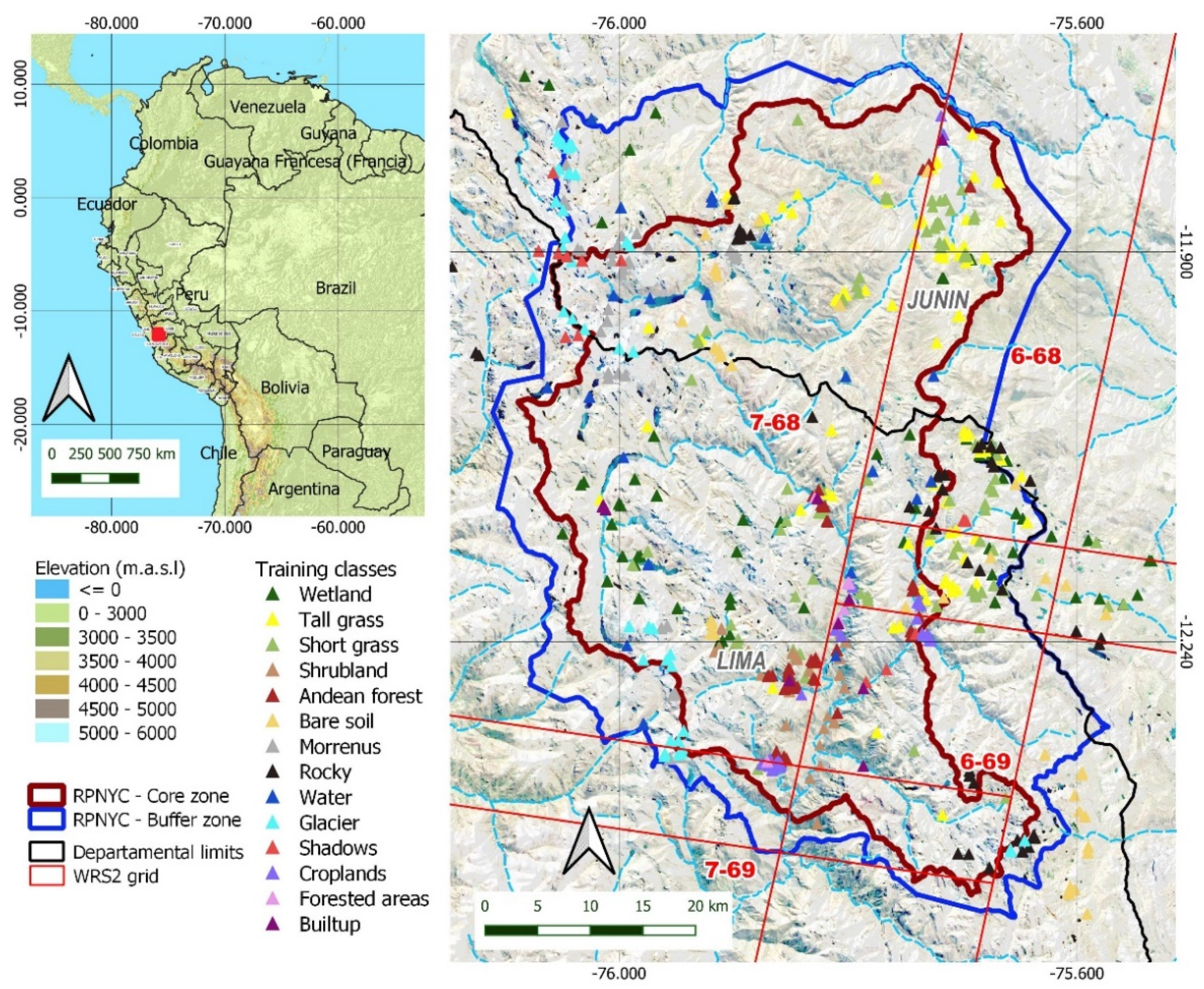
安第斯高原生态系统在地形多样的地区维持着高水平的花卉和动物生物多样性,并提供各种生态系统服务,包括向城市和下游农业谷地供水。谷歌(™)已经开发了一个专门用于测绘的产品(地球引擎),它使用户能够近乎实时地利用基于云计算的解决方案的计算能力,进行土地覆盖变化的测绘和监测。我们探索了在地形复杂、植被类型高度混杂的地形(位于秘鲁安第斯山脉中部的Nor Yauyos Cochas景观保护区)中,利用分类机器学习(ML)算法结合不同的遥感数据,使用这一平台绘制土地覆盖类型的可行性。这些算法使用3601个采样像素进行训练,这些像素包括:(a)2018年期间Landsat 8 OLI传感器的可见光和近红外光谱之间的归一化光谱带;(b)植被、土壤、水、雪、烧毁区和裸地的光谱指数;(c)地形衍生指数(海拔、坡度和坡度)。测试了六种ML算法,包括CART、随机森林、梯度树提升、最小距离、天真贝叶斯和支持向量机。结果显示,当光谱带与地形指数结合使用时,ML算法产生了准确的分类,导致具有类似光谱特征的类别之间更好的区分,如pajonal(草丛为主的覆盖)和短草或岩石群,以及冰碛、农业和森林地区。使用随机森林算法,从光谱带和地形指数的组合中获得了具有最高解释力的模型(Kappa=0.81)。我们的研究提出了在地形复杂的科迪勒拉山脉地形中的第一种方法,我们表明,GEE在快速变化和转换的山区生态系统的大规模土地覆盖制图和监测中特别有用,并可复制和扩展到具有类似特征的其他地区。
方法学框架
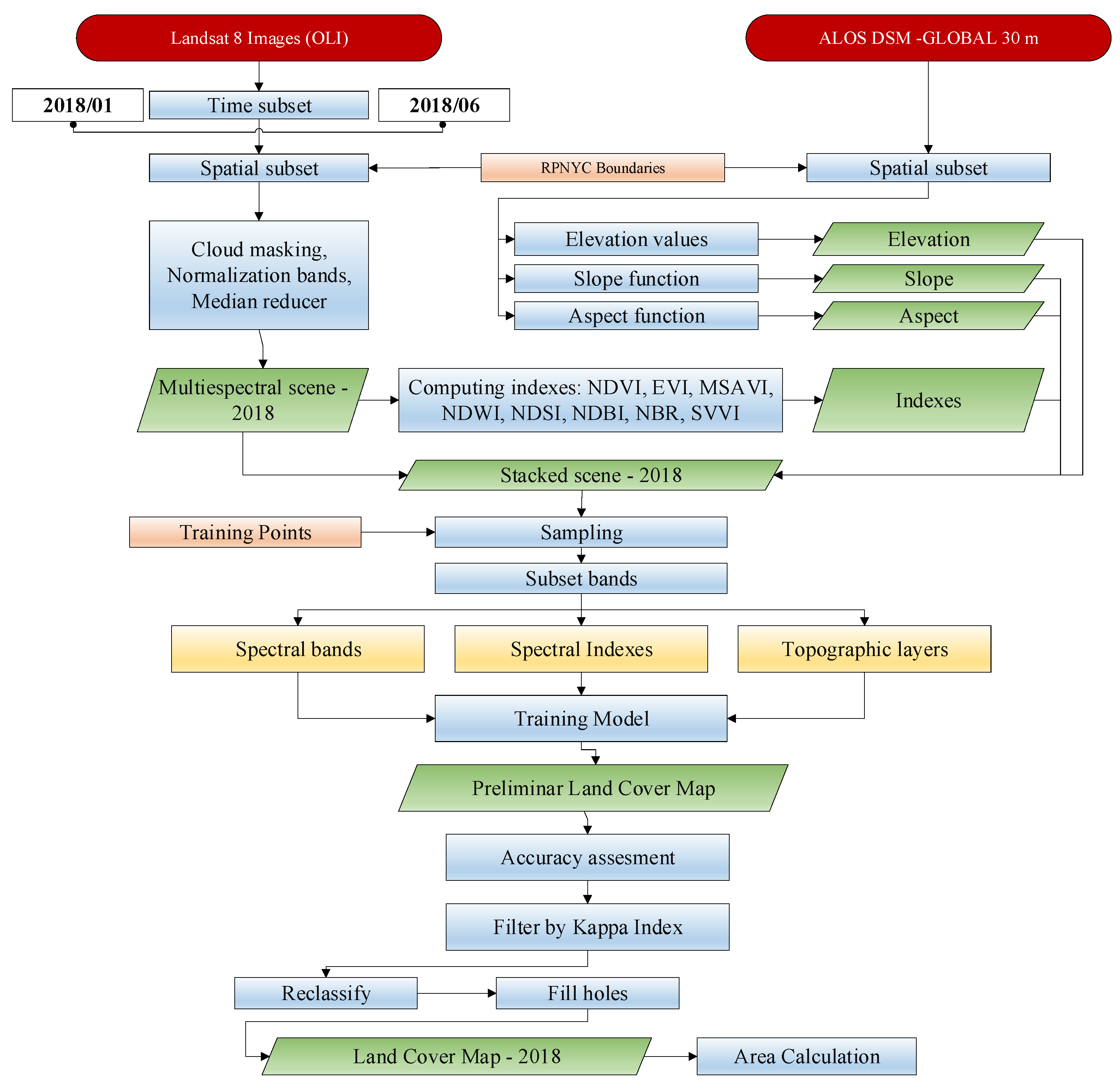
本研究采用的方法学框架见图2,并在以下六个方法分节中描述。Landsat图像预处理(第2.3节)、训练和验证样本收集(第2.4节)、分类特征输入(第2.5节)、可分离性分析(第2.6节)、分类器(第2.7节)、精度评估(第2.8节)和土地覆盖范围的计算(第2.9节)。
整个分类过程:
精度验证:
我们使用R语言中的 "caret"[54]和 "diffeR"[55]包计算了六种计算算法在六组上的混淆矩阵和每个结果分类输出的精度评估。我们计算了总体精度(OA)、Kappa系数(K)、生产者精度(PA)和用户精度(UA),并辅以数量差异(Q)和分配差异(A)评估[56]。我们使用这些指标组合来评估所得到的土地覆盖图的质量。OA决定了一个算法的整体效率,通过正确分类样本的总数除以测试样本的总数来衡量。
K表示地面真实数据和预测值之间的一致程度,而PA衡量一个像素被分类的程度,包括遗漏误差(地面上观察到的特征被错误地排除在一个类别之外的比例)。UA衡量地图的可靠性,说明地图在多大程度上代表了地面上的情况,它包括委托误差,指的是被错误地包含在一个类别中的像素[57]。
Q是指参考数据和对比地图之间由于类别比例不完全匹配而产生的差异量,A是指参考数据和对比地图之间由于类别空间分配不理想而产生的差异量,考虑到参考地图和对比地图中的类别比例(例如,在一个没有观察到建筑发展的地方将建筑分类)[56]。
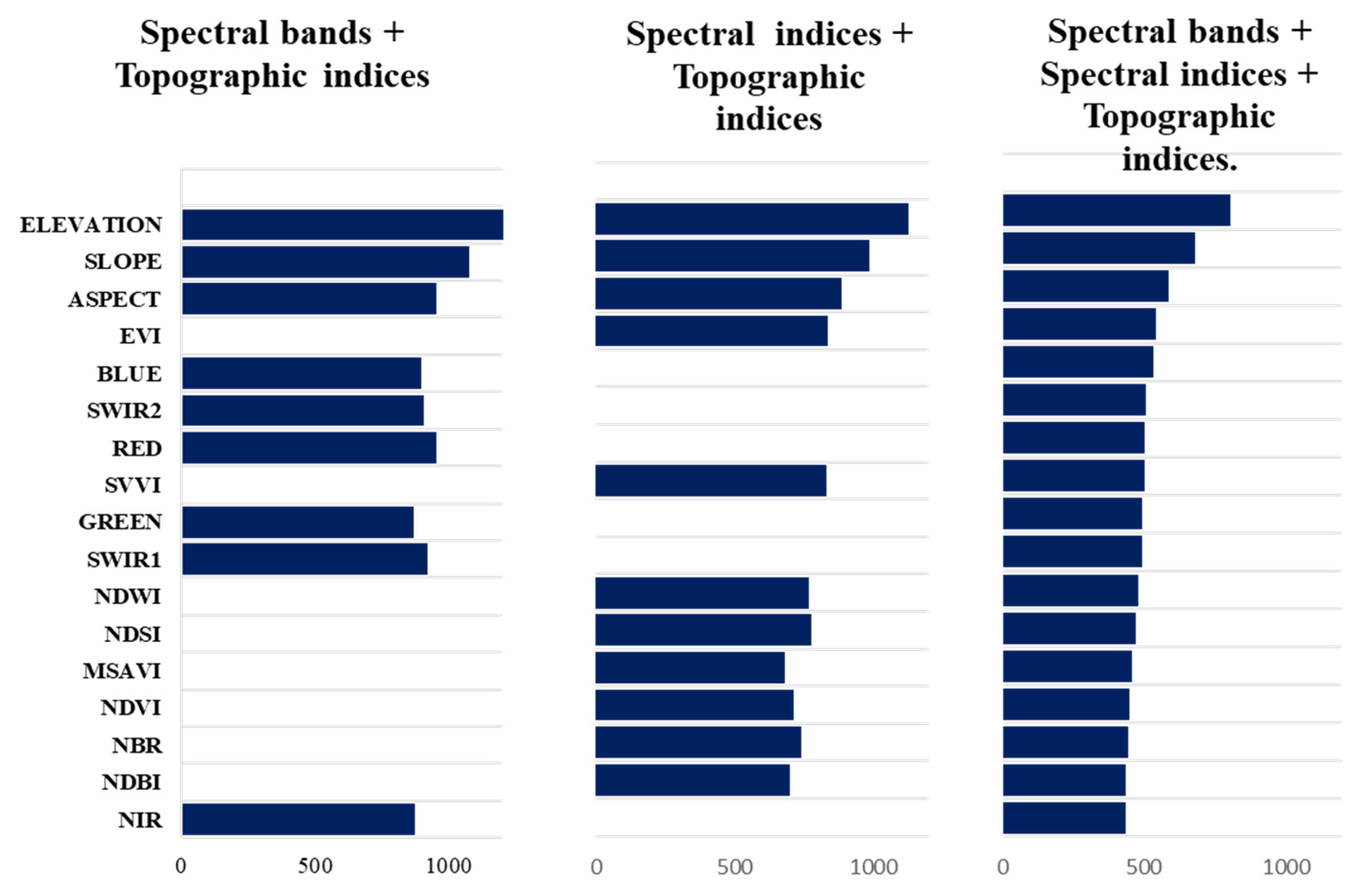
最后,我们使用了变量重要性,它认为每次在一个变量上对节点进行分割时,两个后代节点的不纯度标准要小于父节点,将森林中所有树上的每个单独变量的减少量相加,可以得到一个快速的变量重要性,该重要性通常与包络重要性测量非常一致。这为我们提供了一个额外的手段来评估每个预测变量如何在优化的土地覆盖分类模型中实现准确性的提高,以归一化的百分比贡献。
不同波段组合(变量)产生的分类结果重要性的差异:
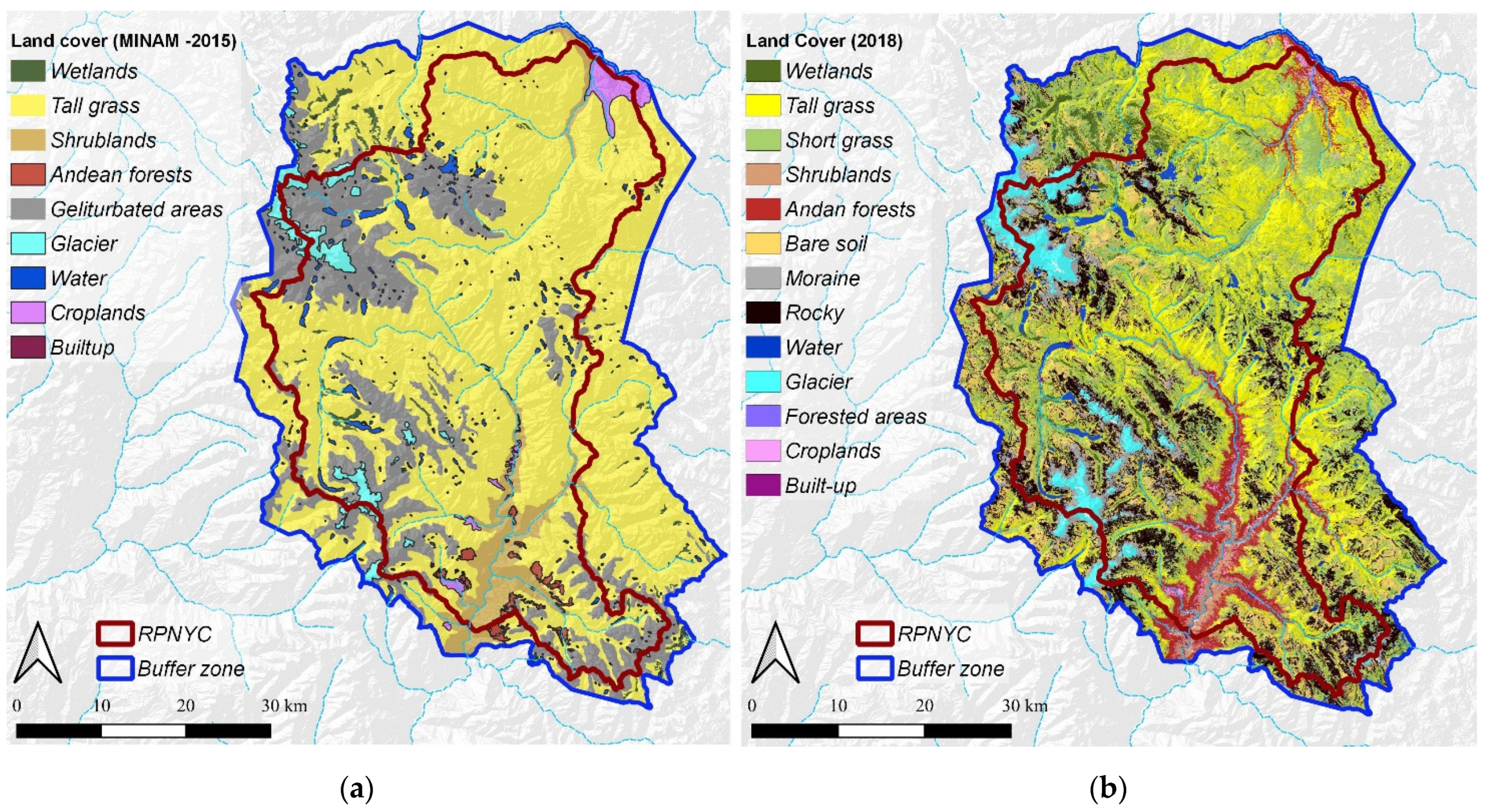
最终的分类结果:
我们的结果表明,使用基于云的计算和机器学习可以使我们建立一个开放的、准确的和相对无差距的方法,利用免费提供的Landsat图像在一个异质的大型安第斯生态系统中绘制土地覆盖地图。这种方法可用于推动欠发达国家的自然资源规划和管理,也有助于通过建立任何参考年份的关键土地覆盖类别,为偏远保护区的植被长期监测提供信息。此外,确定土地覆被等级的范围和分布,以及它们的光谱和地形特征,可以使我们将这些分类推断到类似生态系统中的非保护区域。我们的工作强调了在区分分布在不同的、高度异质的地形中的复杂土地覆盖物时,纳入源于地形的指数的重要性,以提高分类的准确性,这与纳入光谱指数不同。最后,我们建立了一种在最能代表安第斯生态系统的保护区内生成可靠的土地覆盖图的方法,从而为未来的变化分析和环境规划建立了基线。这项工作对实现侧重于监测和评估山区生态系统变化的可持续发展目标15.4以及对长期监测和评估山区环境变化和保护工作具有意义。
这个代码并不能直接使用,因为我们缺少训练样本和测试样本,但是整体的思路就是我们把样本点换成我们自己的就行了。
代码:
//加载研究区样本点信息
var rois = ee.FeatureCollection(train_p1);
var rois2 = ee.FeatureCollection(testing_p1);
//地形参数
var ALOSDSMDEM = ee.Image('JAXA/ALOS/AW3D30_V1_1');
var ALOSDM = ALOSDSMDEM.select('AVE').toDouble().clip (puna).rename("ELEVACION");
var SLOPE_F = ee.Terrain.slope(ALOSDM).toDouble().clip (puna).rename("PENDIENTE");
var ASPECT_F = ee.Terrain.aspect(ALOSDM).toDouble().rename("ASPECTO");
var SIN_ASPECT =
ASPECT_F.divide(180).multiply(Math.PI).sin().toDouble().rename("ASPECTO");
//光学影像
var IMGLandsat8= ee.ImageCollection ('LANDSAT/LC08/C01/T1_SR')
.filterDate ('2018-01-31', '2018-06-30') //fechas disponibles ('2013-04-11' - actualidad)
.filterBounds (puna);
//影像去云
function maskLXsr(image){
var cloudShadowBitMask = ee.Number(2).pow(3).int();
var cloudsBitMask = ee.Number(2).pow(5).int();
var qa = image.select('pixel_qa');
var mask = qa.bitwiseAnd(cloudShadowBitMask).eq(0)
.and(qa.bitwiseAnd(cloudsBitMask).eq(0));
return image.updateMask(mask).divide(10000).clamp(1e-6, 1)
.copyProperties(image, ["system:time_start"]);
}
//影像去云映射和中位数合成
var lXmask = IMGLandsat8.map(maskLXsr);
var Landsat8_NRG1 = ee.Image(lXmask.median());
//波段替换和转化为双精度
var Landsat8a = Landsat8_NRG1.clip (puna)
.select(['B2', 'B3', 'B4', 'B5', 'B6', 'B7'])
.rename(['blue','green','red','nir','swir1','swir2'])
.toDouble();
//
var Landsat8 = (Landsat8a.select('blue','green','red','nir','swir1','swir2')
.divide((Landsat8a.select('blue','green','red','nir','swir1','swir2')
.reduce(ee.Reducer.sum())).divide(ee.Number(6))))
.multiply(ee.Number(100));
//添加指数
//添加 NDVI
var addNDVI=function(imagen){
var NIR='nir';
var RED='red';
var ndvi= imagen.normalizedDifference([NIR, RED]).rename('NDVI');
return imagen.addBands(ndvi).select('NDVI')};
var ndvi = addNDVI(Landsat8).select('NDVI').toDouble().rename("NDVI");
var NDVI_FIN = ndvi.clamp(-1, 1);
//print(NDVI_FIN,"NDVI_FIN")
//Map.addLayer(NDVI_FIN,{min: 0, max: 1},"ndvi");
//添加NDBI
// Normalized Difference Builtup Index (NDBI) NDBI = (SWIR1 – NIR) / (SWIR1 + NIR)
var addNDBI=function(imagen){
var NIR='nir';
var SWIR1='swir1';
var ndbi= imagen.normalizedDifference([SWIR1,NIR]).rename('NDBI');
return imagen.addBands(ndbi).select('NDBI')};
var NDBI = addNDBI(Landsat8).select('NDBI').toDouble().rename("NDBI");
var NDBI_FIN = NDBI.clamp(-1, 1);
//print(NDBI_FIN,"NDBI_FIN")
//Map.addLayer(NDBI_FIN,{min: 0, max: 1},"NDBI");
//添加NBR
//Normalized BURN RATIO "(NIR - SWIR1) / (NIR + SWIR1)"
var addNBR=function(imagen){
var NIR='nir';
var SWIR1='swir1';
var nbr= imagen.normalizedDifference([NIR, SWIR1]).rename('NBR');
return imagen.addBands(nbr).select('NBR')};
var NBR = addNBR(Landsat8).select('NBR').toDouble().rename("NBR");
var NBR_FIN = NBR.clamp(-1, 1);
//print(NBR_FIN,"NBR_FIN")
//Map.addLayer(NBR_FIN,{min: 0, max: 1},"NBR");
//添加NDWI
// Normalized Difference Water Index (NDWI) NDWI = (Green – NIR) / (Green + NIR)
var addNDWI=function(imagen){
var NIR='nir';
var GREEN='green';
var ndwi= imagen.normalizedDifference([GREEN, NIR]).rename('NDWI');
return imagen.addBands(ndwi).select('NDWI')};
var NDWI = addNDWI(Landsat8).select('NDWI').toDouble().rename("NDWI");
var NDWI_FIN = NDWI.clamp(-1, 1);
//print(NDWI_FIN,"NDWI_FIN")
//Map.addLayer(NDWI_FIN,{min: 0, max: 1},"NDWI");
//添加NBR NDSI
// Modified Normalized Difference Water Index (mNDWI) or Normalized Difference Snow Index (NDSI)
// A common index for detecting water (mNDWI) or snow (NDSI), where mNDWI = NDSI = (Green – SWIR1) / (Green + SWIR1).
var addNDSI=function(imagen){
var GREEN='green';
var SWIR1='swir1';
var ndsi= imagen.normalizedDifference([GREEN, SWIR1]).rename('NDSI');
return imagen.addBands(ndsi).select('NDSI')};
var NDSI = addNDSI(Landsat8).select('NDSI').toDouble().rename("NDSI");
var NDSI_FIN = NDSI.clamp(-1, 1);
//print(NDSI_FIN,"NDSI_FIN")
//Map.addLayer(NDSI_FIN,{min: 0, max: 1},"NDSI");
// 添加 MSAVI
var MSAVI = Landsat8.select('nir').multiply(2).add(1)
.subtract(Landsat8.select('nir').multiply(2).add(1).pow(2)
.subtract(Landsat8.select('nir').subtract(Landsat8.select('red')).multiply(8)).sqrt()
).divide(2)
var MSAVI_FIN = MSAVI.toDouble().rename("MSAVI");
//print(MSAVI_FIN,"MSAVI_FIN")
//Map.addLayer(MSAVI_FIN,{min: 0, max: 0.5},"MSAVI_FIN");
// 添加 Enhanced Vegetation Index (EVI)
//An "optimized" vegetation index designed to enhance the vegetation signal with improved
sensitivity
//in high biomass regions. Landsat EVI = 2.5 * (NIR – Red) / ( NIR + 6*Red – 7.5*Blue + 1).
var EVI1 = ((Landsat8.select('nir').subtract(Landsat8.select('red')))
.divide(Landsat8.select('nir').add(((Landsat8.select('red').multiply(6))
.subtract(Landsat8.select('blue').multiply(7.5))).add(1)))).multiply(2.5)
var EVI = EVI1.toDouble().rename("EVI");
//print (EVI, 'EVI')
//Map.addLayer(EVI, {min: -1, max: 1}, 'EVI');
//添加光谱变异植被指数 Spectral Variability Vegetation Index (SVVI)
var SVV1a = Landsat8.select('blue','green','red','nir','swir1','swir2').reduce(ee.Reducer.stdDev());
var SVV1b = Landsat8.select('nir','swir1','swir2').reduce(ee.Reducer.stdDev());
var SVVI = SVV1a.subtract(SVV1b).rename("SVVI");
//var bands = ['blue','green','red','nir','swir1','swir2']
//var bands = ['NDVI','NDBI','NBR', 'NDWI','NDSI','MSAVI','EVI','SVVI'];
//var bands = ['blue','green','red','nir','swir1','swir2', 'NDVI','NDBI','NBR', 'NDWI','NDSI','MSAVI','EVI','SVVI']
//var bands = ['blue','green','red','nir','swir1','swir2','ELEVACION', 'PENDIENTE', 'ASPECTO'];
//var bands = ['NDVI','NDBI','NBR', 'NDWI','NDSI','MSAVI','EVI','SVVI', 'ELEVACION', 'PENDIENTE', 'ASPECTO'];
//var bands = ['blue','green','red','nir','swir1','swir2','NDVI','NDBI','NBR', 'NDWI','NDSI','MSAVI','EVI','SVVI','ELEVACION', 'PENDIENTE', 'ASPECTO'];
//多波段融合
var predictors_base = Landsat8.addBands(NDVI_FIN).addBands(NDBI_FIN).addBands(NBR_FIN)
.addBands(NDWI_FIN).addBands(NDSI_FIN).addBands(MSAVI_FIN)
.addBands(EVI).addBands(SVVI)
.addBands(ALOSDM).addBands(SLOPE_F).addBands(SIN_ASPECT);
//选择不同波段用于训练模型
var predictors = predictors_base.select(bands);
//这里有两个一个是训练样本一个是测试样本
//提取样本点这里直接变成矢量集合
var trainingPartition = predictors.sampleRegions({
collection: rois,
properties: ['Clave'],
scale: 30
});
var testingPartition = predictors.sampleRegions({
collection: rois2,
properties: ['Clave'],
scale: 30
});
//多分类器集合为一个变量 为下一步应用不同模型进行准备
var classifiers = [ee.Classifier.libsvm(),
ee.Classifier.minimumDistance(),
ee.Classifier.smileCart(),
ee.Classifier.smileGradientTreeBoost(100),
ee.Classifier.smileNaiveBayes(),
ee.Classifier.smileRandomForest(100)];
//进行随机森林训练
var classifier = ee.Classifier.smileRandomForest({numberOfTrees: 100,}).train({
features: trainingPartition,
classProperty: 'Clave',
inputProperties: bands
});
//描述经过训练的分类器的结果。
var dict = classifier.explain();
//变量重要性重要性发分析
var variable_importance = ee.Feature(null, ee.Dictionary(dict).get('importance'));
//加载变量重要性图表
var chart =
ui.Chart.feature.byProperty(variable_importance)
.setChartType('ColumnChart')
.setOptions({
title: 'Random Forest Variable Importance',
legend: {position: 'none'},
hAxis: {title: 'Bands'},
vAxis: {title: 'Importance'}
});
var votes = ee.Image().clip(puna);
//这里遍历所有分类方法得出结果
for(var i in classifiers) {
votes = votes.addBands(predictors.classify(classifiers[i].train(trainingPartition,"Clave")))}
print(votes, "votes");
var votes1 = votes.select('classification_4').toDouble()
print(votes1, 'vot1')
//导出分类结果
*Export.image.toDrive({
image: votes.select('classification','classification_1',
'classification_2', 'classification_3',
'classification_4', 'classification_5'),
description: 'C1',
scale: 30,
folder : 'COVER_NYC',
maxPixels: 8000000000,
region: puna});
数据引用:
Pizarro SE, Pricope NG, Vargas-Machuca D, Huanca O, Ñaupari J. Mapping Land Cover Types for Highland Andean Ecosystems in Peru Using Google Earth Engine. Remote Sensing. 2022; 14(7):1562. https://doi.org/10.3390/rs14071562


评论(0)