抗体优化新方法:通过AI预测亲和力和自然度
今天给大家介绍的是由来自Absci公司的Vancouver (WA)团队发表在bioRxiv上的预印文章《Antibody optimization enabled by artificial intelligence predictions of binding affinity and naturalness》。这篇文章提出利用人工智能对抗体进行优化的基于高通量亲和数据训练的深层语境语言模型(deep contextual language models trained on high-throughput affinity data),并运用名为ACE和SPR的方法,用于生成抗体结合亲和力的相对传统方法而言更优的测量,然后基于两种不同的抗体证明了可以定量预测未知抗体序列变体的结合。

ACE方法的提出,解决了传统方法对序列空间探索度较低的问题,从而使得文中提出的深层语境语言模型可以高精度地定量预测未知抗体变体的结合亲和力,实现虚拟筛选,并将可访问序列空间扩大了几个数量级。
研究背景
传统的抗体优化方法仅仅只能探索到序列空间的很小区域,这样的情况下,优化的结果有可能会被限制在仅有次优性质的序列上,这些序列有可能会结合亲和力不足、发育受到限制或者具有较差的免疫原性谱。相比之下,深度诱变加上筛选或选择允许探索更大的抗体序列空间。但是突变本身也具有降低抗体的结合亲和力等问题,这将会大大降低筛选效率。深度神经网络是一种可以有效帮助克服实验筛选能力的限制的工具,但是现有的模型受到吞吐量和二进制(而非连续)读数的限制,这可能会在高突变负载下影响其准确性。
在本研究中,作者使用深层语境语言模型和定量、高通量的实验结合亲和性数据,证明了提高抗体与其目标抗原结合亲和性的能力。经过验证,该模型可以高精度地定量预测未知抗体变体的结合亲和力,实现虚拟筛选,并将可访问序列空间扩大了几个数量级。最后,在实验室中的预测和后续设计可以确认成功率远远高于传统筛选。
主要结果
使用深度语言模型预测序列变体的结合亲和力
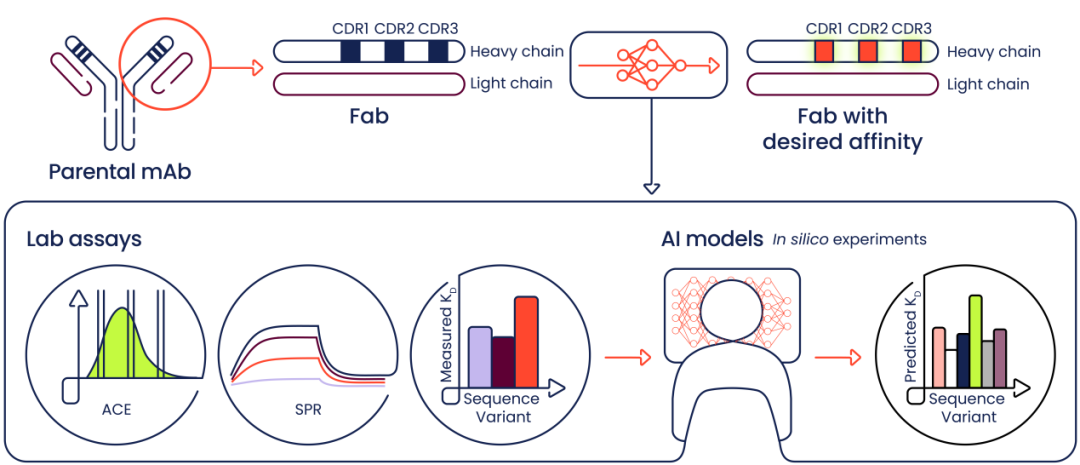 图1 人工智能增强抗体优化示意图
图1 人工智能增强抗体优化示意图
深度学习模型需要高质量的大量数据进行训练,为了产生抗体结合亲和力的高通量测量,作者提出了基于FACS(荧光激活细胞分类术,Fluorescence-Activated Cell Sorting)和NGS(新一代测序技术,Next-Generation Sequencing)的ACE方法。ACE方法的特点是利用折叠抗体的细胞内可溶性过表达,表达抗体变体的细胞被固定、渗透并用荧光标记的抗原和支架靶向探针染色。然后根据结合亲和力和变体的表达水平对细胞进行装箱和分类。最后,收集的DNA序列通过PCR扩增并测序。SPR方法的特点是低通量,但是准确度更高,因此在数据中用作对ACE数据的补充。所使用的深度语言模型都经过了OAS数据库的免疫球蛋白序列训练,最终的结果证明了模型具有预测序列变体的结合亲和力的能力。
表1 训练模型所用数据
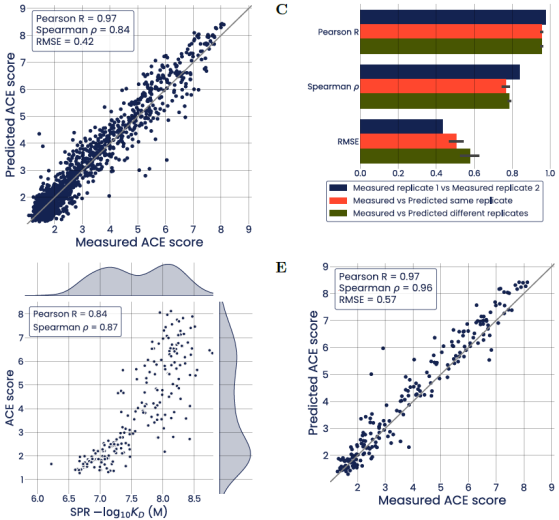
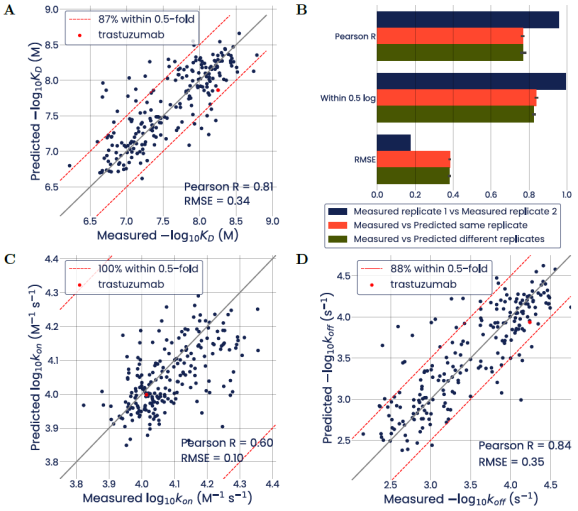 图2 数据集trast-1和trast-2的预测抗体结合亲和力结果
图2 数据集trast-1和trast-2的预测抗体结合亲和力结果
如图1所示,作者使用trast-1数据集对深度语言模型进行训练,并保留10%的数据用于测试。测试的结果表明数据集的ACE得分和模型的预测得分高度相关。为了进一步评估预测性能,trast-2数据集由从trast-1数据集中采样了>200个序列,通过SPR重新筛选100个采样序列,作者证实了该数据集的结合亲和度的接近均匀分布并验证了模型在trast-2的强大预测性能。图2展示了预测抗体结合亲和力结果,其中比较的两个基线分别为未经过预训练的深度语言模型和基于XGBoost梯度增强树。
改进抗体变体的模型引导设计
作者使用模型设计具有期望绑定特性的序列集,然后用专用的SPR实验进行验证。首先在trast-2数据集上训练一个模型,并让它设计50个跨越两个数量级平衡解离常数的序列(设计集A),作者发现设计集A的预测和验证之间非常一致。
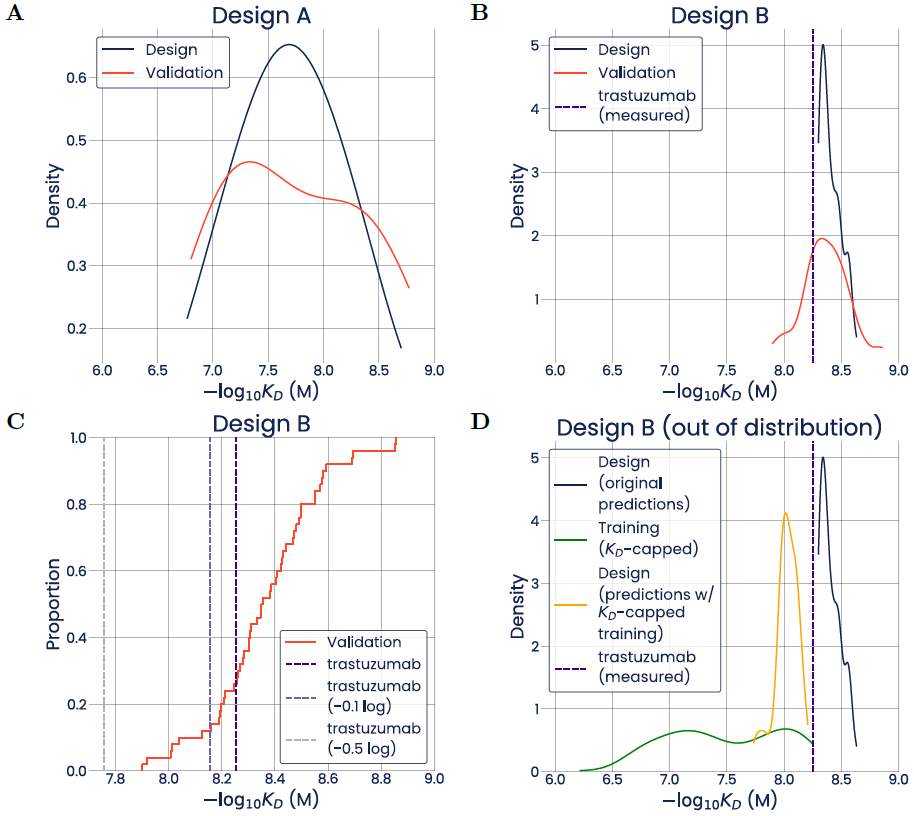 图3 使用trast-2训练的模型上次的设计集A和B与预测值关系图
图3 使用trast-2训练的模型上次的设计集A和B与预测值关系图
然后用该模型进行比曲妥珠单抗结合更紧密的变体的设计(设计集B),与之前相同,让模型设计了50个序列,通过SPR验证这50个序列,发现74%的变体确实比亲本抗体更紧密。100%的变体在0.5log的误差范围内符合设计要求。
设计集B的验证结果与单纯的、仅在实验室进行文库筛选的方法相比有很大的优势,在该方法中,比曲妥珠单抗更紧密的复合物比例最小。因此人工智能模型提供的变体的强富集可以极大地促进抗体优化。
作者注意到,生成设计集B的模型是通过trast-2数据集训练得到的,里面有一些比曲妥珠单抗结合性更强的结合剂。在除去这些结合剂训练后,新得到的模型不再能够为设计做出准确的KD预测。尽管如此,该模型确实将设计B变体的结合亲和度放在其预测分布的顶部。这一结果表明,即使实验室实验产生的训练数据没有跨越完全的亲和度范围,AI也可以实现高亲和度序列的优先级化。
其他的讨论
在后续研究中,作者发现当扩展到更大的序列空间时,AI预测的表现仍然能够保持在不错的水平上,并且作者提出的深度语言模型具有很高的样本效率,使抗体结合的可解释分析成为可能。
在抗体的优化方面,着眼于优化抗体的自然性可以缓解发展障碍。将候选抗体开发为治疗药物是一个复杂的过程,具有高度的临床前和临床风险。由于难以获得信息丰富且相关的数据,对这些风险进行建模一直是行业面临的巨大挑战。作者将自然度定义为通过预先训练的语言模型计算的分数,该分数测量给定抗体序列从感兴趣物种(包括人类)衍生的可能性。因此,自然度可以用作抗体设计和工程中的指导指标。
总结
本文在之前的相关模型基础上提出了运用人工智能进行抗体优化的思路,并引入抗体的自然度定义,提出SPR和ACE方法,大大提高了序列空间的探索度,为人工智能模型提供了足够优质的高通量数据,最终人工智能模型设计得到的序列展现出了很强的亲和力和稳定性,并且在扩展到更大的序列空间时仍然具有很好的预测水平。
参考资料
https://www.biorxiv.org/content/10.1101/2022.08.16.504181v1
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/126495803
- 点赞
- 收藏
- 关注作者


评论(0)