全球生物量 2003-2019 数据集提供了地球陆地木本植被生态系统的地上生物量存量和变化的估计值
概述
Chloris Global Biomass 2003 - 2019 数据集提供了地球陆地木本植被生态系统的地上生物量存量和变化的估计值。它涵盖了 2003 年至 2019 年期间,按年度时间步长计算。全球数据集的空间分辨率约为 4.6 公里。
这些地图和数据集是通过结合来自星载卫星的多个遥感测量结果生成的,使用最先进的机器学习和统计方法进行处理,并使用来自多个国家的现场数据进行验证。该数据集提供了对地上储量和变化的直接估计,而不是基于土地利用或土地覆盖面积的变化,因此它们包括所有类型的木本植被(无论是自然植被还是人工林植被)中碳储量的收益和损失。
年度存量以生物量吨数为单位。库的年度变化以二氧化碳当量单位表示,即该特定像素从陆地生态系统释放或吸收的二氧化碳量。
空间数据集可在上使用 Attribution-Non Commercial-Share Alike 类型的 Creative Common 许可。
是一家以使命为导向的技术公司,开发有关自然资本状况的软件和数据产品,供企业、政府和社会部门使用。
STAC 集合
提供者
(生产商、许可人)(主机、处理器)执照
空间范围
地图样式:grayscale_light。
时间范围
2003 年 7 月 31 日 – 2019 年 7 月 31 日GSD
4633 米项目级资产
数据集项目包含以下资产。
biomass
biomass_wm
biomass_change
biomass_change_wm
数据集资产
abfs://items/chloris-biomass.parquet
geoparquet-items
Chloris 全球生物量 2003-2019 数据集提供了地球陆地木本植被生态系统的地上生物量存量和变化的估计值。它涵盖了 2003-2019 年期间,按年度时间步长计算。全球数据集的空间分辨率约为 4.6 公里。
此笔记本提供了使用 Planetary Computer STAC API 访问 Chloris Biomass 数据、检查目录中的数据资产以及对来自 Cloud Optimized GeoTIFF 源的数据进行一些简单处理和绘图的示例。
环境设置
此笔记本可使用或不使用 API 密钥,但您将获得使用 API 密钥访问数据的更多权限。Planetary Computer Hub 已预先配置为使用您的 API 密钥。
环境设定
查询可用数据
返回的17件物品
我们的搜索返回了该系列的所有17个项目。每个项目都代表了一个单一的年份,在2003年至2019年之间,在全球范围内。
让我们看看有哪些资产与这个项目有关。
chloris_biomass_50km_2019:
- biomass: Annual estimates of aboveground woody biomass.
- biomass_wm: Annual estimates of aboveground woody biomass (Web Mercator).
- biomass_change: Annual estimates of changes (gains and losses) in aboveground woody biomass from the previous year.
- biomass_change_wm: Annual estimates of changes (gains and losses) in aboveground woody biomass from the previous year (Web Mercator).
- tilejson: TileJSON with default rendering
- rendered_preview: Rendered preview有4项资产,尽管有两项是相同数据的重复,以Web Mercator投影方式提供。其中一个代表了给定年份的地上木质生物量的估计值(吨),另一个代表了与前一年相比的变化(吨)。
让我们更新我们的搜索,只包括一个特定的年份。
加载感兴趣的变量
通过检查每个资产的额外字段中的raster:band数组,我们可以看到这个数据集的 "nodata "值是2,147,483,647。所以我们将为rioxarray提供masked=True选项,以打开数据,将nodata转换为NaN。
xarray.Dataset
- Dimensions:
- y: 3600
- x: 8640
- Coordinates: (3)
-
x
(x)
float64
-2.001e+07 -2.001e+07 ... 2.001e+07
-
y
(y)
float64
1.001e+07 1e+07 ... -6.669e+06
-
spatial_ref
()
int64
0
-
- Data variables: (1)
-
biomass
(y, x)
float64
...
-
- Attributes: (2)
scale_factor :
1.0
add_offset :
0.0
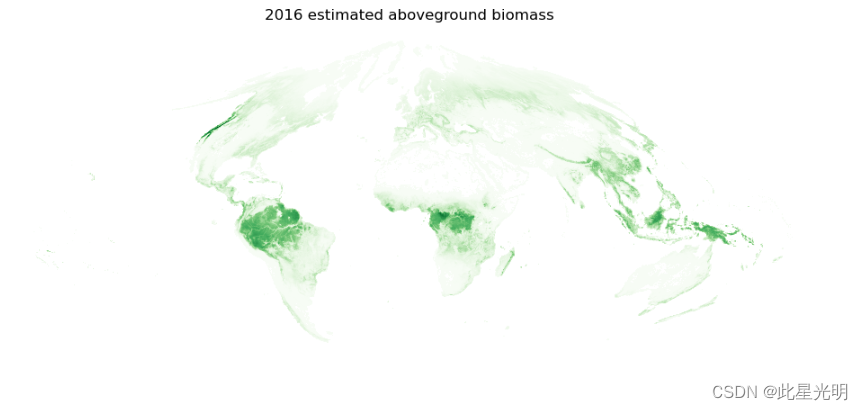
对于这个全局图,在我们的渲染中损失一些细节是可以的。首先,我们将在每个空间维度上对整个数据集进行10倍的降样,并将节点数据值以上的任何数值删除。
CPU times: user 697 ms, sys: 270 ms, total: 967 ms
Wall time: 1.14 s随着我们的数据集被很好地缩小,我们可以在原始的正弦投影中绘制地球的地面生物量图
- 点赞
- 收藏
- 关注作者


评论(0)