全球生物量数据产品(python)
概述
全球(GBIF) 是一个由世界各国政府资助的国际网络和数据基础设施,提供记录物种发生的全球数据。GBIF 目前整合了记录超过 16 亿种物种出现的数据集。
GBIF 发生数据集结合了来自广泛来源的数据,包括来自自然历史博物馆的标本相关数据、来自公民科学网络的观察和自动环境调查。虽然这些数据在上不断变化,但会在此处拍摄并提供定期快照。
数据以格式存储;Parquet 文件架构如下所述。大多数字段名称对应于,并且已经被 GBIF 的系统解释为对齐分类、位置、日期等。可以使用检索附加信息。
有关如何在出版物中引用 GBIF 数据的信息,请参阅 GBIF。对于使用整个数据集的分析,请使用以下引用:
GBIF.org ([Date]) GBIF Occurrence Data [数据集的 DOI]
对于数据被显着过滤的分析,请跟踪使用的 datasetKeys 并使用“”记录来引用数据。
包含许多可以帮助您分析 GBIF 数据的文章。
STAC 集合
提供者
(生产者、许可者、加工者)(主持人)执照
空间范围
Map panned east 8515.94 kilometers.
Map style: grayscale_light.
时间范围
2021 年 4 月 13 日 – 至今项目级资产
数据集项目包含以下资产。
data
帐户名称
ai4edataeuwest列
每个表包括以下列。
Columns
Each table includes the following columns.
Name
Description
Type
Gbifid
GBIF’s identifier for the occurrence
Int64
Datasetkey
GBIF’s UUID for the containing this occurrence
Byte_array
Occurrenceid
See .
Byte_array
Kingdom
See . This field has been aligned with the .
Byte_array
Phylum
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Class
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Order
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Family
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Genus
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Species
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Infraspecificepithet
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Taxonrank
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Scientificname
See . This field has been aligned with the GBIF backbone taxonomy.
Byte_array
Verbatimscientificname
The scientific name as provided by the data publisher
Byte_array
Verbatimscientificnameauthorship
The scientific name authorship provided by the data publisher.
Byte_array
Countrycode
See . GBIF’s interpretation has set this to an ISO 3166-2 code.
Byte_array
Locality
See .
Byte_array
Stateprovince
See .
Byte_array
Occurrencestatus
See . Either the value PRESENT or ABSENT. Many users will wish to filter for PRESENT data.
Byte_array
Individualcount
See .
Int32
Publishingorgkey
GBIF’s UUID for the publishing this occurrence.
Byte_array
Decimallatitude
See . GBIF’s interpretation has normalized this to a WGS84 coordinate.
Double
Decimallongitude
See . GBIF’s interpretation has normalized this to a WGS84 coordinate.
Double
Coordinateuncertaintyinmeters
See .
Double
Coordinateprecision
See .
Double
Elevation
See . If provided by the data publisher, GBIF’s interpretation has normalized this value to metres.
Double
Elevationaccuracy
See . If provided by the data publisher, GBIF’s interpretation has normalized this value to metres.
Double
Depth
See . If provided by the data publisher, GBIF’s interpretation has normalized this value to metres.
Double
Depthaccuracy
See . If provided by the data publisher, GBIF’s interpretation has normalized this value to metres.
Double
Eventdate
See . GBIF’s interpretation has normalized this value to an ISO 8601 date with a local time.
Byte_array
Day
See .
Int32
Month
See .
Int32
Year
See .
Int32
Taxonkey
The numeric identifier for the in GBIF’s backbone taxonomy corresponding to scientificname.
Int32
Specieskey
The numeric identifier for the taxon in GBIF’s backbone taxonomy corresponding to species.
Int32
Basisofrecord
See . One of PRESERVED_SPECIMEN, FOSSIL_SPECIMEN, LIVING_SPECIMEN, OBSERVATION, HUMAN_OBSERVATION, MACHINE_OBSERVATION, MATERIAL_SAMPLE, LITERATURE, UNKNOWN.
Byte_array
Institutioncode
See .
Byte_array
Collectioncode
See .
Byte_array
Catalognumber
See .
Byte_array
Recordnumber
See .
Byte_array
Identifiedby
See .
Byte_array
Dateidentified
See . An ISO 8601 date.
Byte_array
License
See . Either or . CC_BY_NC_4_0 records are not present in this snapshot.
Byte_array
Rightsholder
See .
Byte_array
Recordedby
See .
Byte_array
Typestatus
See .
Byte_array
Establishmentmeans
See .
Byte_array
Lastinterpreted
The ISO 8601 date when the record was last processed by GBIF. Data are reprocessed for several reasons, including changes to the backbone taxonomy, so this date is not necessarily the date the occurrence record last changed.
Byte_array
Mediatype
See . May contain StillImage, MovingImage or Sound (from , detailing whether the occurrence has this media available.
Byte_array
Issue
A list of encountered by GBIF in processing this record. More details are available on these issues and flags in .
Byte_array
PRESENT或ABSENT。 许多用户希望过滤PRESENT数据。
scientificname.
species.
PRESERVED_SPECIMEN, FOSSIL_SPECIMEN, LIVING_SPECIMEN, OBSERVATION, HUMAN_OBSERVATION, MACHINE_OBSERVATION, MATERIAL_SAMPLE,LITERATURE之一UNKNOWN。
CC_BY_NC_4_0此快照中不存在记录。
StillImage, MovingImageor Sound(from , 详细说明事件是否有此媒体可用。
数据集资产
abfs://items/gbif.parquet
geoparquet-items
使用 Planetary Computer STAC API 访问 GBIF 数据
此笔记本提供了从 Planetary Computer STAC API访问数据的定期快照以 Parquet 格式存储。
我们将使用读取分区 Parquet 数据集。
['gbif-2021-09-01',
'gbif-2021-08-01',
'gbif-2021-07-01',
'gbif-2021-06-01',
'gbif-2021-04-13']我们将采取最新的项目。
<项目id=gbif-2021-09-01>。
像往常一样,你应该在尝试加载数据之前签署该项目。
Dask DataFrame Structure:
| gbifid | datasetkey | occurrenceid | kingdom | phylum | class | order | family | genus | species | infraspecificepithet | taxonrank | scientificname | verbatimscientificname | verbatimscientificnameauthorship | countrycode | locality | stateprovince | occurrencestatus | individualcount | publishingorgkey | decimallatitude | decimallongitude | coordinateuncertaintyinmeters | coordinateprecision | elevation | elevationaccuracy | depth | depthaccuracy | eventdate | day | month | year | taxonkey | specieskey | basisofrecord | institutioncode | collectioncode | catalognumber | recordnumber | identifiedby | dateidentified | license | rightsholder | recordedby | typestatus | establishmentmeans | lastinterpreted | mediatype | issue | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| npartitions=1034 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| int64 | object | object | object | object | object | object | object | object | object | object | object | object | object | object | object | object | object | object | int32 | object | float64 | float64 | float64 | float64 | float64 | float64 | float64 | float64 | object | int32 | int32 | int32 | int32 | int32 | object | object | object | object | object | object | object | object | object | object | object | object | object | object | object | |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
Dask Name: read-parquet, 1034 tasks
正如npartitions所表明的,这个Parquet数据集是由许多单独的parquet文件组成的。我们可以用.get_partition读入一个特定的partition
| gbifid | datasetkey | occurrenceid | kingdom | phylum | class | order | family | genus | species | ... | identifiedby | dateidentified | license | rightsholder | recordedby | typestatus | establishmentmeans | lastinterpreted | mediatype | issue | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 321355870 | 67fb29a4-f762-11e1-a439-00145eb45e9a | None | Chromista | Foraminifera | Globothalamea | Rotaliida | Globigerinidae | Globigerina | Globigerina bulloides | ... | None | None | CC_BY_4_0 | None | Schiebel, Ralf | None | None | 2021-06-14T23:58:35.923Z | [] | [] |
| 1 | 321355956 | 67fb29a4-f762-11e1-a439-00145eb45e9a | None | Chromista | Foraminifera | Globothalamea | Rotaliida | Globorotaliidae | Neogloboquadrina | Neogloboquadrina pachyderma | ... | None | None | CC_BY_4_0 | None | Schiebel, Ralf | None | None | 2021-06-14T23:58:35.924Z | [] | [] |
| 2 | 321355949 | 67fb29a4-f762-11e1-a439-00145eb45e9a | None | Chromista | Foraminifera | Globothalamea | Rotaliida | Globigerinitidae | Globigerinita | Globigerinita minuta | ... | None | None | CC_BY_4_0 | None | Schiebel, Ralf | None | None | 2021-06-14T23:58:35.926Z | [] | [] |
| 3 | 321355912 | 67fb29a4-f762-11e1-a439-00145eb45e9a | None | Chromista | Foraminifera | Globothalamea | Rotaliida | Globigerinitidae | Globigerinita | Globigerinita glutinata | ... | None | None | CC_BY_4_0 | None | Schiebel, Ralf | None | None | 2021-06-14T23:58:35.927Z | [] | [] |
| 4 | 321355905 | 67fb29a4-f762-11e1-a439-00145eb45e9a | None | Chromista | Foraminifera | Globothalamea | Rotaliida | Globigerinidae | Globigerina | Globigerina falconensis | ... | None | None | CC_BY_4_0 | None | Schiebel, Ralf | None | None | 2021-06-14T23:58:35.928Z | [] | [] |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 1667009 | 1322591709 | 821cc27a-e3bb-4bc5-ac34-89ada245069d | http://n2t.net/ark:/65665/3ee636308-1443-45be-... | Animalia | Arthropoda | Malacostraca | Decapoda | Processidae | Processa | Processa bermudensis | ... | Mclaughlin, P. | None | CC0_1_0 | None | Continental Shelf Associates for BLM/ MMS | None | None | 2021-06-27T10:49:36.284Z | [StillImage] | [OCCURRENCE_STATUS_INFERRED_FROM_INDIVIDUAL_CO... |
| 1667010 | 1322595622 | 821cc27a-e3bb-4bc5-ac34-89ada245069d | http://n2t.net/ark:/65665/3ee8fc491-fa05-4edc-... | Animalia | Chordata | Mammalia | Rodentia | Muridae | Hybomys | Hybomys univittatus | ... | None | None | CC0_1_0 | None | J. Malcolm | None | None | 2021-06-27T10:50:21.046Z | [StillImage] | [OCCURRENCE_STATUS_INFERRED_FROM_INDIVIDUAL_CO... |
| 1667011 | 1322597346 | 821cc27a-e3bb-4bc5-ac34-89ada245069d | http://n2t.net/ark:/65665/3eea331a2-d225-4e88-... | Animalia | Chordata | Amphibia | Caudata | Plethodontidae | Plethodon | Plethodon montanus | ... | None | None | CC0_1_0 | None | None | None | None | 2021-06-27T10:49:36.308Z | [] | [OCCURRENCE_STATUS_INFERRED_FROM_INDIVIDUAL_CO... |
| 1667012 | 1322605552 | 821cc27a-e3bb-4bc5-ac34-89ada245069d | http://n2t.net/ark:/65665/3eeff553c-637b-4742-... | Animalia | Chordata | Thaliacea | Salpida | Salpidae | Salpa | None | ... | Cole, Linda L., (IZ), Smithsonian Institution ... | None | CC0_1_0 | None | Schuyler | None | None | 2021-06-27T10:50:57.439Z | [] | [OCCURRENCE_STATUS_INFERRED_FROM_INDIVIDUAL_CO... |
| 1667013 | 1456448120 | 821cc27a-e3bb-4bc5-ac34-89ada245069d | http://n2t.net/ark:/65665/3ef2a984e-e0aa-4544-... | Plantae | Tracheophyta | Magnoliopsida | Fabales | Fabaceae | Swartzia | Swartzia polyphylla | ... | Torke, B. M., (MO) | None | CC0_1_0 | None | R. Oldeman | None | None | 2021-06-27T10:49:36.344Z | [StillImage] | [OCCURRENCE_STATUS_INFERRED_FROM_INDIVIDUAL_CO... |
1667014 rows × 50 columns
为了了解最常观察到的物种,我们将对数据集进行分组,并获得每个物种的计数。
kingdom phylum class family genus species
Animalia Chordata Aves Sulidae Sula Sula variegata 143568
Actinopterygii Percidae Perca Perca fluviatilis 48700
Cyprinidae Rutilus Rutilus rutilus 39743
Esocidae Esox Esox lucius 37786
Percidae Gymnocephalus Gymnocephalus cernua 23030
Cyprinidae Abramis Abramis brama 17225
Lotidae Lota Lota lota 15668
Cyprinidae Alburnus Alburnus alburnus 14580
Salmonidae Coregonus Coregonus maraena 14381
Coregonus albula 13104
Mammalia Phocoenidae Phocoena Phocoena phocoena 12250
Actinopterygii Osmeridae Osmerus Osmerus eperlanus 11782
Salmonidae Salmo Salmo trutta 10384
Salvelinus Salvelinus alpinus 10340
Cyprinidae Scardinius Scardinius erythrophthalmus 9120
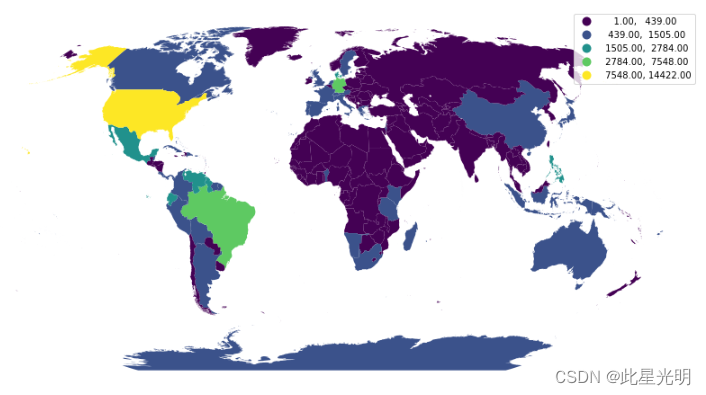
Name: species, dtype: int64让我们用每个国家的独特物种数量创建一个地图。首先,我们将按国家代码分组,并计算出独特物种的数量
countrycode
AD 6
AE 55
AF 32
AG 51
AI 37
...
YE 19
YT 5
ZA 688
ZM 82
ZW 91
Name: species, Length: 最后,我们可以通过将species_per_country连接到有国家边界的数据集上,用geopandas在地图上绘制计数。243, dtype: int64
使用完整的数据集
到目前为止,我们只是使用了整个 GBIF 数据集中的一个分区。本手册中的所有例子都是在整个数据集上使用dask.dataframe来读取Parquet数据集的。
你可能想创建一个集群,在许多机器上并行处理数据。
现在你可以重复上面的计算,用df代替chunk。
往期推荐:
Google Earth Engine(GEE)——Sentinel-2数据你真的用对了吗?
Google Earth Engine(GEE)—— GRIDMET: 爱达荷大学网格化地表气象数据集
Google Earth Engine——全球陆地冰层空间数据的介绍(内含常见错误)
利用GEE-APP(LT-GEE Time Series Animator)快速实现1984-2021年gif动图的下载
Google Earth Engine APP——不写代码实现影像图例添加和全球底图添加
Google Earth Engine APP——不写代码实现影像图例添加和全球底图添加
- 点赞
- 收藏
- 关注作者


评论(0)