设备管理中设备名称聚类分析
简 介: 本文对于设备名称进行了分词聚类,并对聚类结果中仪器所使用的机时进行了腹部统计。通过对词典长度参数, SOM 聚类 边长参数进行调整,查看参数对于聚类结果的影响,找到最合理的参数。通过测试可以看到 SOM 聚类边长为 10×10 时,网络聚类效果最好。词典不同的长度对于聚类分析结果影响不大。通过对聚类结果所使用机时进行直方图统计,可以看到: 大部分的分布还是呈现于总体分布相似的情况,指数分布,这说明对于名称聚类对机时分布统计没有太多的影响。 对于少部分的聚类结果,分布不在呈现指数分布,但机时分布方差更大;综上所述,可以看到仅仅通过对设备名称进行聚类,对于设备使用机时分布没有能够得到更加合理的划分。
关键词: 设备,分类,聚类,机时,使用分布,直方图

§01 设备名称聚类
一、背景介绍
为了对于设备使用时间进行归类评价,需要建立设备正常使用的分布标准。在 设备管理中数据聚类处理-预处理 对来自 JYH 的数据进行了初步的分析,提取所有设备的名称,并进行分词统计。下面的工作包括:
- 建立起适当的分词词典,对设备名称表示成词典向量;
- 对设备进行聚类。
二、词典建立
在 设备管理中数据聚类处理-预处理 中对于所有设备名称进行了分词统计,获得了 2306 个分词,他们具有不同的出现频次。 下面给出了出现频次最多的前 20 个分词。
[['系统' '2347']
['仪' '989']
['分析仪' '900']
['显微镜' '744']
['激光' '619']
['激光器' '516']
['测试' '329']
['扫描' '319']
['质谱' '295']
['机' '255']
['光谱仪' '247']
['分析' '223']
['联用' '216']
['荧光' '212']
['探针' '188']
['平台' '186']
['装置' '185']
['聚焦' '177']
['色谱' '173']
['真空' '173']]
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
1、确定词典长度
先按照单词出现频次进行排序,选择其中 前 N 项 组成字典,对所有 11531 条记录中的设备名称进行查询,能够匹配上至少一条词典词语的称为匹配名称。 统计词典长度与匹配数据之间的曲线,如下图所示。可以看到选择前 200 ~ 300 条词语形成词典,便可以覆盖 98% 以上的记录。

▲ 图1.2.1 词典长度与匹配覆盖率
为了使得设备名称维度不太大, 先设定词典的长度为 200 ,建立设备名称的向量数据。 下面是词典中的词语:
['系统', '仪', '分析仪', '显微镜', '激光', '激光器', '测试', '扫描', '质谱', '机']
['光谱仪', '分析', '联用', '荧光', '探针', '平台', '装置', '聚焦', '色谱', '真空']
['谱仪', '成像', '高速', 'X射线', '信号', '共', '测量', '网络', '液相', '质谱仪']
['电子显微镜', '测试仪', '高', '纯化', '设备', '飞秒', '原子力', '沉积', '矢量', '流式细胞']
['台', '试验机', '采集', '低温', '热', '多功能', '发射', '显微', '离子', '刻蚀']
['红外', '三维', '与', '半导体', '衍射', '探测器', '相色谱', '分辨', '蛋白', '场']
['等离子体', '光学', '快速', '发生器', '检测', '气', '光纤', '全自动', '拉曼', '精密']
['脉冲', '数据', '超', '热仪', '红外光谱仪', '-', '动态', '实验', '试验台', '示波器']
['超高', '扫描电镜', '工作站', '气体', '多', '光谱', '处理', '控制系统', '纳米', '电子束']
['高温', '微波', '自动', '倒置', '等离子', '频谱', '摄像机', '分子', '及', '炉']
['飞行', '微量', '任意', '/', '无', '试验', '耦合', '核磁共振', '电', '微']
['高性能', '数字', '变换', '高压', '测量仪', '加工', '细胞', '超速离心', '粒子', '干涉仪']
['摩擦', '仿真', '放大器', '波形发生器', '离子束', '杆', '隧道', '超声', '实验台', '定量']
['透射', '冷冻', '光', '等温', '高效', '化学', '同步', '吸附', '生物', '镀膜']
['双', '相机', '制冷机', '滴定', '光度计', '振动', '能谱仪', '流变', '蒸发', '型']
['实时', '高纯锗', '高功率', '时间', '综合', '扫描仪', 'PCR', '样机', '气相', '跟踪']
['环境', '表面', '控制器', '磁控溅射', '合成', '集群', '在线', '光子', '式', '颗粒物']
['反应', '中心', '阻抗', '烧结炉', '液氦', '原子', '傅里叶', '模拟', '粒径', '制备']
['器', '信号源', '样品', '分光', '四级', '电池', '伺服', '光电子', '便携式', '计算']
['晶体', '稀释', '分选', '全', '高分辨率', '3D', '钛', '电子', '光谱分析', '图像']
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
2、设备名称向量
利用上述 200 个单词组成了词典,对于所有设备的名称建立向量数据。向量的长度为 200。 对于11531 条记录中的设备名称,便于10465 个记录设备名称中向量不全为 0。
下面给出了两条仪器名称记录所对应的数据向量。
三室`真空`定向`炉`
[0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]
光电`发射`光谱仪
[0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
三、名称聚类
在 sklearn-som 中给出了 sklearn 中的自组织特征映射(SOM)聚类方法的使用。
1、初步测试
(1)名称聚类
初步构造一个 10×10 的 SOM 网络, 对于11531 个记录中的名字进行聚类。
from headm import *
from sklearn_som.som import SOM
namevect = tspload('namevect200', 'namevect')
SOM_SIDE = 10
VECT_LEN = shape(namevect)[1]
name_som = SOM(m=SOM_SIDE,n=SOM_SIDE,dim=200)
name_som.fit(namevect)
predictions = name_som.predict(namevect)
tspsave('prediction', pred=predictions)
printf('\a')
printf(predictions)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
下面是聚类后,每一个 SOM 神经元对应的样本个数。通过观察,可以看到其中包括有六个主要的聚类节点,它们对应的样本总数为 4600, 占总数的 40% 左右。
000 000 000 000 000 000 000 000 000 000
000 000 304 077 000 098 084 043 000 000
160 179 101 034 000 446 005 079 000 000
009 000 008 197 119 000 142 053 033 154
384 049 140 000 170 116 183 085 000 076
159 000 110 033 025 103 158 212 083 159
000 130 189 040 060 241 2000 259 000 120
253 028 007 094 000 115 110 000 980 154
000 000 486 000 103 111 000 127 196 190
172 046 178 107 000 029 079 016 226 115
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10

▲ 图1.3.1 对应的样本出现频次以及所形成的六个主要的聚类节点
(2)聚类结果分析
Ⅱ.聚类名称
下面给出了聚类命中个数最多的前 6 个对应的一起名称中前 10 个仪器名称。
第1类,个数:2000
1:直流电源及其控制系统
2:无人驾驶电动飞艇
3:氦气风机
4:自然循环大模拟实验
5:氦净化工程验证回路
6:气相色谱仪
7:气相色谱仪
8:超声多普勒测速仪
9:粒径速度测定仪
10:立式氦气压缩机
第2类,个数:980
1:摇摆综合模拟台架系统
2:DR-CT安全检查系统
3:转筒式固态发酵罐系统
4:单倾冷冻传输杆系统
5:超高灵敏度检漏系统
6:光纤光谱测量系统
7:光学成像系统
8:显微摄像系统
9:晶体培养观察系统
10:蛋白质结晶系统
第3类,个数:486
1:高级扩展流变仪
2:化学吸附仪
3:实时高速喷雾粒度仪
4:蛋白纯化仪
5:全自动磁性细胞分选仪
6:激光光斑跟踪仪
7:台阶仪
8:显微注射仪
9:流变仪
10:形貌仪
第4类,个数:446
1:酶联免疫斑点分析仪
2:自动采样颗粒物分析仪
3:气溶胶粒径分析仪
4:个体化基因分析仪
5:动态力学分析仪
6:比表面积及孔隙分析仪
7:模态分析仪
8:模态测量分析仪
9:连续流动分析仪
10:总磷总氮分析仪
第5类,个数:384
1:高分辨率磁力显微镜
2:原子力显微镜
3:高级荧光显微镜
4:电动倒置显微镜
5:显微镜
6:原子力显微镜
7:荧光显微镜
8:倒置生物显微镜
9:显微镜操作系统
10:原子力显微镜
第6类,个数:304
1:氩离子激光器
2:脉冲准分子激光器
3:固体激光器
4:激光器
5:氪离子激光器
6:准分子激光器
7:固体激光器
8:脉冲激光器
9:染料激光器
10:红宝石激光器
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
Ⅲ.聚类机时分布
统计上面六个主要的聚类中心,对应的使用机时分布。 下面给出了利用仪器名称聚类所对应的设备分布直方图。
- 聚类前 6 类的仪器有效使用时间分布直方图。

▲ 对应聚类使用时间直方图
- 聚类排名 7 ~ 12 的仪器使用时间直方图分布:

▲ 对应聚类设备使用时间直方图
2、改变参数测试
(1)改变 SOM 尺寸
下面是将 SOM 的边长 改变为 15×15,对应的聚类结果。可以看到SOM中存在大量的 0 区域,这说明 SOM 变大对于聚类效果没有改善。

▲ SOM边长为15×15对应的聚类结果

▲ 对应聚类前6所使用机时分布图
下面是 SOM 边长为 5×5 时对应的聚类结果。
247 294 159 490 900
090 007 266 456 000
591 153 615 2900 605
005 000 352 074 000
199 795 001 529 1803
- 1
- 2
- 3
- 4
- 5

▲ SOM边长为 5×5对应的聚类结果

▲ 聚类前6类对应的使用机时分布直方图
通过以上对于尺寸的变化,可以看到,实际上二维 SOM的尺寸在 10×10 左右最佳,此时,SOM中的大部分神经元都被调动起来。对于 SOM 聚类对应的使用机时分布来看,大部分还是呈现指数分布。 在尺寸为 15×15 的时候, 出现的更大范围的随机分布。
(2)改变词典长度
下面给出词典长度分别为 100,150,200,250,300 时,对于 SOM 的尺寸为 10×10 时聚类结果,以及前 6 个聚类对应的机时使用时间分布。
Ⅱ.词典长度为100

▲ 聚类分布

▲ 前六类机时直方图
Ⅲ.词典长度为150

▲ SOM聚类结果

▲ 前6类使用机时分布图
Ⅳ.词典长度为200

▲ 聚类结果

▲ 聚类前6类使用机时分布直方图
Ⅴ.词典长度为250

▲ 聚类结果
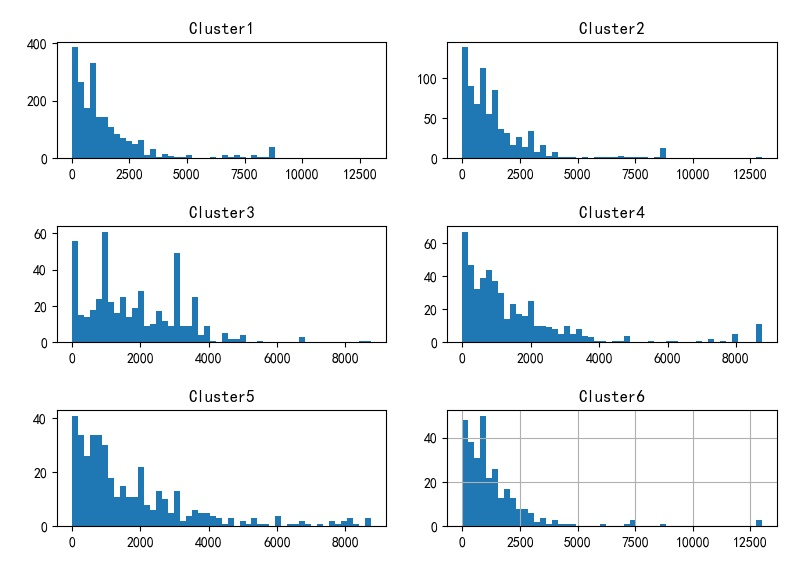
▲ 聚类前6类使用机时分布直方图
Ⅵ.词典长度为300

▲ 聚类结果

▲ 聚类结果前6类使用时间直方图
※ 总 结 ※
本文对于设备名称进行了分词聚类,并对聚类结果中仪器所使用的机时进行了腹部统计。通过对词典长度参数, SOM 聚类 边长参数进行调整,查看参数对于聚类结果的影响,找到最合理的参数。通过测试可以看到 SOM 聚类边长为 10×10 时,网络聚类效果最好。词典不同的长度对于聚类分析结果影响不大。
通过对聚类结果所使用机时进行直方图统计,可以看到:
- 大部分的分布还是呈现于总体分布相似的情况,指数分布,这说明对于名称聚类对机时分布统计没有太多的影响。
- 对于少部分的聚类结果,分布不在呈现指数分布,但机时分布方差更大;
综上所述,可以看到仅仅通过对设备名称进行聚类,对于设备使用机时分布没有能够得到更加合理的划分。
■ 相关文献链接:
● 相关图表链接:
- 图1.2.1 词典长度与匹配覆盖率
- 图1.3.1 对应的样本出现频次以及所形成的六个主要的聚类节点
- 对应聚类使用时间直方图
- 对应聚类设备使用时间直方图
- SOM边长为15×15对应的聚类结果
- 对应聚类前6所使用机时分布图
- SOM边长为 5×5对应的聚类结果
- 聚类前6类对应的使用机时分布直方图
- 聚类分布
- 前六类机时直方图
- SOM聚类结果
- 前6类使用机时分布图
- 聚类结果
- 聚类前6类使用机时分布直方图
- 聚类结果
- 聚类前6类使用机时分布直方图
- 聚类结果
- 聚类结果前6类使用时间直方图
from headm import *
cutname = tspload('namecut', 'cutname')
wordall = tspload('word', 'wordall')
dictlen = 300
dictdim = [w[0] for w in wordall[:dictlen]]
namevect = []
matchnum = 0
for id,n in enumerate(cutname):
nn = n.split('`')
nv = []
flag = 0
for d in dictdim:
if d in nn:
nv.append(1)
flag = 1
else:
nv.append(0)
namevect.append(nv)
matchnum += flag
if id % 100 == 0: printf('%d:%d'%(id, matchnum))
printf(matchnum, len(namevect))
tspsave('namevect300', namevect=namevect)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
#!/usr/local/bin/python
# -*- coding: gbk -*-
#============================================================
# CLUSTANALY.PY -- by Dr. ZhuoQing 2022-08-11
#
# Note:
#============================================================
from headm import *
pred = tspload('prediction', 'pred')
cutname = tspload('namecut', 'cutname')
attrstr, alldata = tspload('alldata', 'attrstr', 'alldata')
#------------------------------------------------------------
alltime = array([float(a[3]) for a in alldata])
predlist = list(pred)
neuralcount = [predlist.count(i) for i in range(100)]
sortcount = sorted(zip(neuralcount, list(range(len(neuralcount)))), key=lambda x:x[0], reverse=True)
#------------------------------------------------------------
for i in range(6):
namelist = cutname[pred==sortcount[i][1]]
printf(' ')
printf("第%d类,个数:%d"%(i+1, len(namelist)))
for id,n in enumerate(namelist[:10]):
printf("%d:%s"%(id+1, n.replace('`','')))
#------------------------------------------------------------
startid = 0
for i in range(6):
timelist = alltime[pred==sortcount[i+startid][1]]
plt.subplot(3,2,i+1)
plt.hist(timelist, bins=50)
plt.title('Cluster%d'%(i+1+startid))
plt.grid(True)
plt.tight_layout()
plt.show()
#------------------------------------------------------------
# END OF FILE : CLUSTANALY.PY
#============================================================
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
#!/usr/local/bin/python
# -*- coding: gbk -*-
#============================================================
# SHOWNAMEVECT.PY -- by Dr. ZhuoQing 2022-08-11
#
# Note:
#============================================================
from headm import *
from sklearn_som.som import SOM
#cutname = tspload('namecut', 'cutname')
namevect = tspload('namevect300', 'namevect')
SOM_SIDE = 10
VECT_LEN = shape(namevect)[1]
name_som = SOM(m=SOM_SIDE,n=SOM_SIDE,dim=VECT_LEN)
name_som.fit(namevect)
predictions = name_som.predict(namevect)
tspsave('prediction', pred=predictions)
printf('\a')
printf(predictions)
#------------------------------------------------------------
# END OF FILE : SHOWNAMEVECT.PY
#============================================================
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
#!/usr/local/bin/python
# -*- coding: gbk -*-
#============================================================
# RESULT.PY -- by Dr. ZhuoQing 2022-08-11
#
# Note:
#============================================================
from headm import *
pred = tspload('prediction', 'pred')
#------------------------------------------------------------
predlist = list(pred)
SOM_SIDE = 10
neuralcount = [predlist.count(i) for i in range(SOM_SIDE**2)]
printf(neuralcount, sum(neuralcount))
countstr = ['%03d'%n for n in neuralcount]
countstr10 = [' '.join(countstr[i*SOM_SIDE:i*SOM_SIDE+SOM_SIDE]) for i in range(SOM_SIDE)]
showdim = '\n'.join(countstr10)
printf(showdim)
count = array(neuralcount)
#------------------------------------------------------------
sortcount = sorted(neuralcount, reverse=True)
printf(sortcount)
printf(sum(sortcount[:6])/sum(sortcount))
#------------------------------------------------------------
plt.imshow(count.reshape(SOM_SIDE,SOM_SIDE))
plt.show()
#------------------------------------------------------------
# END OF FILE : RESULT.PY
#============================================================
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
#!/usr/local/bin/python
# -*- coding: gbk -*-
#============================================================
# CLUSTANALY.PY -- by Dr. ZhuoQing 2022-08-11
#
# Note:
#============================================================
from headm import *
pred = tspload('prediction', 'pred')
cutname = tspload('namecut', 'cutname')
attrstr, alldata = tspload('alldata', 'attrstr', 'alldata')
#------------------------------------------------------------
alltime = array([float(a[3]) for a in alldata])
predlist = list(pred)
neuralcount = [predlist.count(i) for i in range(100)]
sortcount = sorted(zip(neuralcount, list(range(len(neuralcount)))), key=lambda x:x[0], reverse=True)
#------------------------------------------------------------
for i in range(6):
namelist = cutname[pred==sortcount[i][1]]
printf(' ')
printf("第%d类,个数:%d"%(i+1, len(namelist)))
for id,n in enumerate(namelist[:10]):
printf("%d:%s"%(id+1, n.replace('`','')))
#------------------------------------------------------------
startid = 0
for i in range(6):
timelist = alltime[pred==sortcount[i+startid][1]]
plt.subplot(3,2,i+1)
plt.hist(timelist, bins=50)
plt.title('Cluster%d'%(i+1+startid))
plt.grid(True)
plt.tight_layout()
plt.show()
#------------------------------------------------------------
# END OF FILE : CLUSTANALY.PY
#============================================================
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
文章来源: zhuoqing.blog.csdn.net,作者:卓晴,版权归原作者所有,如需转载,请联系作者。
原文链接:zhuoqing.blog.csdn.net/article/details/126280720
- 点赞
- 收藏
- 关注作者




评论(0)