【缺陷检测】基于matlab形态学水果蔬菜缺陷检测【含Matlab源码 820期】
一、获取代码方式
获取代码方式1:
完整代码已上传我的资源: 【缺陷检测】基于matlab形态学水果蔬菜缺陷检测【含Matlab源码 820期】
获取代码方式2:
通过订阅紫极神光博客付费专栏,凭支付凭证,私信博主,可获得此代码。
备注:订阅紫极神光博客付费专栏,可免费获得1份代码(有效期为订阅日起,三天内有效);
二、形态学缺陷检测简介
1 图像分析及预处理
拍摄图像会产生随机的扰动,图像有一定的噪声,为消除掉图像中的无关信息,对图像进行预处理。
1.1 灰度化
为降低运算量,需要将拍摄的三通道的RGB图像转化为单通道的灰度图像。采用加权平均法的灰度化方法,其中心理学灰度公式根据人眼对RGB三色的敏感程度选择不同的权重:
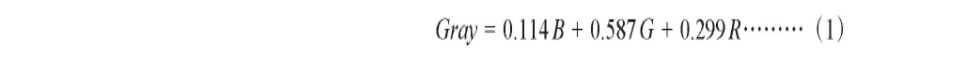
式(1)中,R、G、B分别为RGB三通道灰度值,灰度化结果如图1 (a)所示。
1.2 平滑处理
为了尽可能避免将背景当作缺陷,需要对图像进行平滑处理,这样虽然会使缺陷的边界模糊,但是有利于减少背景的干扰。注意所采用的去噪处理为均值滤波,均值滤波公式为:
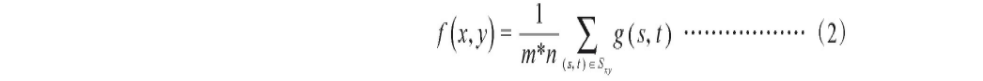
式(2)中,m、n分别为所选择的滤波核的长和宽,Sxy是以(x,y)为中心的滤波核对应像素的位置集合,平滑处理结果如图1 (b)所示。均值滤波的缺点是会使一些细节如边缘等信息丢失,因此在找到种子点后,对没有经过平滑处理的图像进行区域生长,找到缺陷边界。
2 算法原理
2.1 阈值分割
阈值分割是图像分割中最简单、基础的方法,性能比较稳定,计算量较小,运算速度快;它主要有全局阈值分割、局部阈值分割、自适应阈值分割等方法。阈值算法基于阈值T,将像素灰度值大于阈值T和小于阈值T的部分分别叫做前景和背景。变换函数表达式:

图1 均值滤波处理
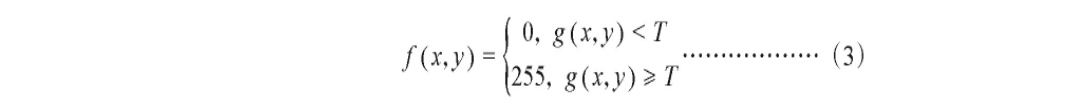
式(3)中,T为阈值,g (x,y)为原图像像素点(x,y)的灰度值,f (x,y)为分割后图像像素点(x,y)的灰度值,阈值分割结果如图2所示。

图2 阈值分割结果
2.2 形态学开运算降噪
数学形态学简称形态学,其处理方式为领域运算,即把领域结构元素与图像对应位置像素进行逻辑运算,这种运算的影响因素主要有结构元素大小、形状和逻辑运算的规则。形态学操作主要有膨胀、腐蚀、梯度运算、礼帽运算、黑帽运算、开运算和闭运算等,但其基础为腐蚀和膨胀,利用膨胀和腐蚀就能完成不同形式的运算。
腐蚀运算能消除轮廓边界点,使边界向内缩小,主要用于细化二值图像目标轮廓、去除噪声等。
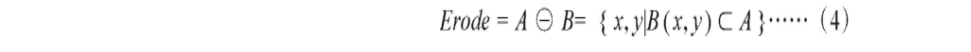
式(4)中,A为原图像,B为结构元素。首先给结构元素B定义一个原点,当结构元素B的原点移动到图像A的(x,y)上时,如果结构元素B上等于1的像素点对应图像A也等于1,则将图像A的(x,y)的灰度值置为1,否则置为0,腐蚀示意图如图3所示。

图3 腐蚀示意图
膨胀运算则与腐蚀运算相反,使边界向外扩张,主要用于填补图像分割后的空白,使相近的不相连的轮廓相连。其公式为:
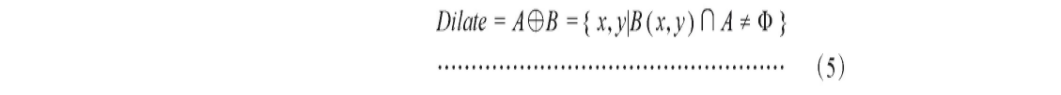
式(5)中,A为原图像,B为结构元素。首先给结构元素B定义一个原点,当结构元素B的原点移动到图像A的(x,y)上时,如果结构元素B上等于1的像素点对应图像A中至少有一个等于1,则将图像A的(x,y)的灰度值置为1,否则置为0。
先进行腐蚀操作,然后在腐蚀的基础上进行膨胀操作,主要用于去噪和计数等。其公式为:
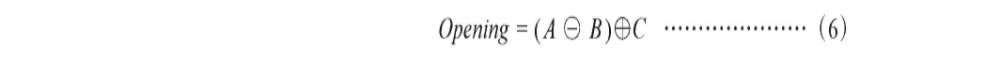
式(6)中,A为原图像,B、C为结构元素。开运算效果如图4所示,图5为开运算处理的结果。

图4 开运算效果
2.3 区域生长法
区域生长的思想就是把领域(四领域、八领域等)相同的化为一个区域。首先需要一个种子点作为生长的开始,然后将种子点领域内满足相似准则要求的像素点合并到种子的区域,将这个区域的像素做为种子点,继续进行生长,直到没有符合要求的点,生长结束,所有种子点像素作为生长的区域。分割的好坏由初始种子点和相似准则决定。

图5 形态学开运算结果
2.3.1 种子点选择与检测
经过阈值分割和形态学处理后,将二值图像各轮廓中心作为待定种子点。如果选择的种子点位于缺陷的绝对区域,那么种子点总有一个方向各像素的深度值呈现高-低-高的形态。设计检测模板如图6所示,计算出种子点在0°、45°、90°、135°方向上的深度变化,判断其变化是否呈高低高形态。
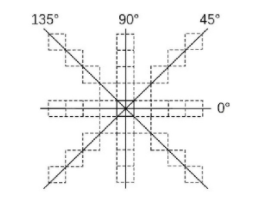
图6 检测模板
种子点左右两侧r个像素的灰度平均值分别为:
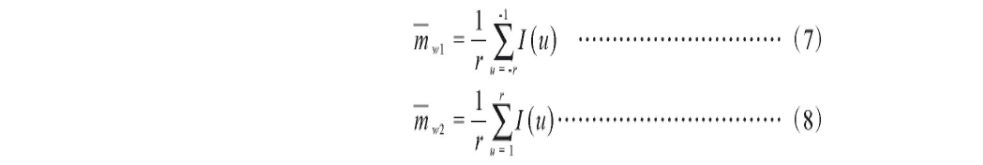
各方向的灰度变化为:
深度形Si态变化判定:
式(10)中,I (u)为检测模板中第u个像素的灰度值,w=1,2,3,4,分别代表0°,45°,90°,135°方向,mwm为w方向两侧的最小灰度值,T1为形态变化阈值。如果种子点不满足深度形态变化判定,则去除该待定种子点。
2.3.2 生长过程
区域生长的具体流程如下:
(1)将种子点坐标放入种子点集seeds。
(2)顶出种子点集中的一个种子点,对种子点八邻域的像素点进行相似准则判断;满足相似准则条件的点,视为种子点放入种子点集seeds。
(3)将顶出的种子点存入种子集S。
(4)如果种子点集内没有元素,则跳到步骤(4);如果种子点集中还有元素,则跳到步骤(2)。
(5)生成一张和输入图像长宽一致,像素值全为0的图像I。
(6)将图像I中对应种子集S坐标的像素值置为255,得到分割图像I’。
其中生长的相似准则为:
式(11)中,gray (seed)为当轮种子点的灰度值,gray (8_n)为其八邻域各点的像素值,Thresh为设置的阈值。区域生长结果如图7所示。

图7 区域生长结果
3 实验过程
图像分割就是按照预先设定的规则,将图像分割为有意义的前景和背景的过程。区域生长是一个分割效果比较好的算法,但前提是需要找到适合的种子点。单一的分割算法就容易遇到这种不足的情况,结合使用形态学和阈值分割的方法来找到合适的种子点,帮助区域生长算法完成分割任务,达到满足要求的分割效果,分割方法流程如图8所示。

图8 分割流程图
首先对输入的图片进行灰度化处理,变成单通道的灰度图片,然后滤波去除噪声,使图像更平滑,选择合适的阈值进行阈值分割,在利用开运算去除掉分割后较小的前景,以各前景区域的中心点为起始种子点,进行区域生长,得到最终所满足要求的前景。
三、部分源代码
ai = imread('a5.jpg');
mi=imread('mango_infected.png');
m4=imread('m4.PNG');
img = imread('potato.png');
type = 'a';
[L, Centers] = imsegkmeans(img, 3);
B = labeloverlay(img,L);
rgbImage = B;
% Display the original image.
subplot(4, 5, 1);
imshow(rgbImage);
title('Original Image(Potato)');
% Split the original image into color bands.
redBand = rgbImage(:,:, 1);
greenBand = rgbImage(:,:, 2);
blueBand = rgbImage(:,:, 3);
% Threshold each color band.
redthreshold = 100;
greenThreshold = 140;
blueThreshold = 170;
redMask = (redBand < redthreshold);
greenMask = (greenBand > greenThreshold);
blueMask = (blueBand < blueThreshold);
% Combine the masks to find where all 3 are 'true.'
damagedAreasMask = uint8(redMask & greenMask & blueMask);
subplot(4, 5, 2);
imshow(damagedAreasMask, []);
title('Damaged Areas Mask');
maskedrgbImage = uint8(zeros(size(damagedAreasMask))); % Initialize
maskedrgbImage(:,:,1) = rgbImage(:,:,1) .* damagedAreasMask;
maskedrgbImage(:,:,2) = rgbImage(:,:,2) .* damagedAreasMask;
maskedrgbImage(:,:,3) = rgbImage(:,:,3) .* damagedAreasMask;
reqInfo = maskedrgbImage(:, :, 1) > 0 & maskedrgbImage(:, :, 2) > 0 & maskedrgbImage(:, :, 3) > 0;
res = sum(reqInfo(:));
disp(res)
t = 10000;
if type=='b'
t = 5000;
end
if res>t
subplot(4, 5, 3);
imshow(maskedrgbImage);
title('Result: Infected');
else
subplot(4, 5, 3);
imshow(maskedrgbImage);
title('Result: Good');
end
%=================================================================================%
img = imread('good_potato.jpg');
[L, Centers] = imsegkmeans(img, 3);
B = labeloverlay(img,L);
rgbImage = B;
% Display the original image.
subplot(4, 5, 4);
imshow(rgbImage);
title('Original Image(Potato)');
% Split the original image into color bands.
redBand = rgbImage(:,:, 1);
greenBand = rgbImage(:,:, 2);
blueBand = rgbImage(:,:, 3);
% Threshold each color band.
redthreshold = 100;
greenThreshold = 140;
blueThreshold = 170;
redMask = (redBand < redthreshold);
greenMask = (greenBand > greenThreshold);
blueMask = (blueBand < blueThreshold);
% Combine the masks to find where all 3 are 'true.'
damagedAreasMask = uint8(redMask & greenMask & blueMask);
maskedrgbImage = uint8(zeros(size(damagedAreasMask))); % Initialize
maskedrgbImage(:,:,1) = rgbImage(:,:,1) .* damagedAreasMask;
maskedrgbImage(:,:,2) = rgbImage(:,:,2) .* damagedAreasMask;
maskedrgbImage(:,:,3) = rgbImage(:,:,3) .* damagedAreasMask;
%imtool(maskedrgbImage);
reqInfo = maskedrgbImage(:, :, 1) > 0 & maskedrgbImage(:, :, 2) > 0 & maskedrgbImage(:, :, 3) > 0;
res = sum(reqInfo(:));
disp(res)
t = 10000;
if type=='b'
t = 5000;
end
if res>t
subplot(4, 5, 5);
imshow(maskedrgbImage);
title('Result: Infected');
else
subplot(4, 5, 5);
imshow(maskedrgbImage);
title('Result: Good');
end
%===============================================================================%
img = imread('banana.png');
%img = imread('b1.jpg');
%img = imread('b2.jpg');
type = 'b';
[L, Centers] = imsegkmeans(img, 3);
B = labeloverlay(img,L);
rgbImage = B;
% Display the original image.
subplot(4, 5, 6);
imshow(rgbImage);
title('Original Image(Banana)');
% Split the original image into color bands.
redBand = rgbImage(:,:, 1);
greenBand = rgbImage(:,:, 2);
blueBand = rgbImage(:,:, 3);
% Threshold each color band.
redthreshold = 100;
greenThreshold = 140;
blueThreshold = 170;
redMask = (redBand < redthreshold);
greenMask = (greenBand > greenThreshold);
blueMask = (blueBand < blueThreshold);
% Combine the masks to find where all 3 are 'true.'
damagedAreasMask = uint8(redMask & greenMask & blueMask);
subplot(4, 5, 7);
imshow(damagedAreasMask, []);
title('Damaged Areas Mask');
maskedrgbImage = uint8(zeros(size(damagedAreasMask))); % Initialize
maskedrgbImage(:,:,1) = rgbImage(:,:,1) .* damagedAreasMask;
maskedrgbImage(:,:,2) = rgbImage(:,:,2) .* damagedAreasMask;
maskedrgbImage(:,:,3) = rgbImage(:,:,3) .* damagedAreasMask;
%imtool(maskedrgbImage);
reqInfo = maskedrgbImage(:, :, 1) > 0 & maskedrgbImage(:, :, 2) > 0 & maskedrgbImage(:, :, 3) > 0;
res = sum(reqInfo(:));
disp(res)
t = 10000;
if type=='b'
t = 5000;
end
if res>t
subplot(4, 5, 8);
imshow(maskedrgbImage);
title('Result: Infected');
else
subplot(4, 5, 8);
imshow(maskedrgbImage);
title('Result: Good');
end
%=================================================================================%
%img = imread('b1.jpg');
img = imread('b2.jpg');
[L, Centers] = imsegkmeans(img, 3);
B = labeloverlay(img,L);
rgbImage = B;
% Display the original image.
subplot(4, 5, 9);
imshow(rgbImage);
title('Original Image(Banana)');
% Split the original image into color bands.
redBand = rgbImage(:,:, 1);
greenBand = rgbImage(:,:, 2);
blueBand = rgbImage(:,:, 3);
% Threshold each color band.
redthreshold = 100;
greenThreshold = 140;
blueThreshold = 170;
redMask = (redBand < redthreshold);
greenMask = (greenBand > greenThreshold);
blueMask = (blueBand < blueThreshold);
% Combine the masks to find where all 3 are 'true.'
damagedAreasMask = uint8(redMask & greenMask & blueMask);
maskedrgbImage = uint8(zeros(size(damagedAreasMask))); % Initialize
maskedrgbImage(:,:,1) = rgbImage(:,:,1) .* damagedAreasMask;
maskedrgbImage(:,:,2) = rgbImage(:,:,2) .* damagedAreasMask;
maskedrgbImage(:,:,3) = rgbImage(:,:,3) .* damagedAreasMask;
%imtool(maskedrgbImage);
reqInfo = maskedrgbImage(:, :, 1) > 0 & maskedrgbImage(:, :, 2) > 0 & maskedrgbImage(:, :, 3) > 0;
res = sum(reqInfo(:));
disp(res)
t = 10000;
if type=='b'
t = 5000;
end
if res>t
subplot(4, 5, 10);
imshow(maskedrgbImage);
title('Result: Infected');
else
subplot(4, 5, 10);
imshow(maskedrgbImage);
title('Result: Good');
end
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
- 77
- 78
- 79
- 80
- 81
- 82
- 83
- 84
- 85
- 86
- 87
- 88
- 89
- 90
- 91
- 92
- 93
- 94
- 95
- 96
- 97
- 98
- 99
- 100
- 101
- 102
- 103
- 104
- 105
- 106
- 107
- 108
- 109
- 110
- 111
- 112
- 113
- 114
- 115
- 116
- 117
- 118
- 119
- 120
- 121
- 122
- 123
- 124
- 125
- 126
- 127
- 128
- 129
- 130
- 131
- 132
- 133
- 134
- 135
- 136
- 137
- 138
- 139
- 140
- 141
- 142
- 143
- 144
- 145
- 146
- 147
- 148
- 149
- 150
- 151
- 152
- 153
- 154
- 155
- 156
- 157
- 158
- 159
- 160
- 161
- 162
- 163
- 164
- 165
- 166
- 167
- 168
- 169
- 170
- 171
- 172
- 173
- 174
- 175
- 176
- 177
- 178
- 179
- 180
- 181
- 182
- 183
- 184
- 185
- 186
- 187
- 188
- 189
- 190
- 191
四、运行结果

五、matlab版本及参考文献
1 matlab版本
2019版
2 参考文献
[1] 蔡利梅.MATLAB图像处理——理论、算法与实例分析[M].清华大学出版社,2020.
[2]杨丹,赵海滨,龙哲.MATLAB图像处理实例详解[M].清华大学出版社,2013.
[3]周品.MATLAB图像处理与图形用户界面设计[M].清华大学出版社,2013.
[4]刘成龙.精通MATLAB图像处理[M].清华大学出版社,2015.
文章来源: qq912100926.blog.csdn.net,作者:海神之光,版权归原作者所有,如需转载,请联系作者。
原文链接:qq912100926.blog.csdn.net/article/details/116104058
- 点赞
- 收藏
- 关注作者


评论(0)