R 主成分与因子分析
【摘要】 主成分与因子分析
@TOC
1 概念
**主成分分析(PCA)**是一种数据降维技巧,它能
将大量相关变量转化为一组很少的不相关变量,
这些无关变量称为主成分。
**因子分析(EFA)**是一用来发现一组
变量的潜在结构的方法。它通过寻找一组更小的、
潜在的或隐藏的结构来解释已观测到的、显式的
变量间的关系。
2 数学模型


不同之处:
主成分分析中的主成分个数与原始变量个数是一样
的,即有几个变量就有几个主成分,只不过最后我
们确定了少数几个主成分而已。 因子分析需要事先确定要找几个成分(因子,
factor),然后将原始变量综合为少数的几个因子,
以再现原始变量与因子之间的关系。一般来说,因
子的个数会远远少于原始变量的个数。 因变量和因子个数的不一致,使得不仅在数学模型上、
而且在实际求解过程中,因子分析和主成分分析都有着
一定的区别,因子分析计算更为复杂



特征值和特征向量

3 相关性分析
读取数据
library(openxlsx)
data0=read.xlsx("2.xlsx",na.strings = "")
data1=data0[,c(-1,-10)]
colclasses=sapply(data0,class)
str(data0)
观察特征值和协方差矩阵的关系
#x协方差矩阵Ʃ对角线上的元素为各变量方差
mycov=round(cov(data1),2)
#求特征值和特征向量
myeign=eigen(mycov)
myvector=myeign$vectors[,1]
mycov%*%myvector
myeign$values[1]*myvector
sum(myeign$values)
sum(diag(mycov))
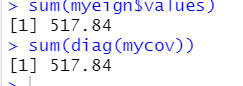
发现协方差矩阵对角线之和等于特征向量之和
#变量名处理,取前三位
name3=substr(names(data0),1,3)
name3=toupper((name3))
rownames(data0)=paste("bank",1:nrow(data0),sep="")
data1=data0[,c(-1,-6,-10)]
target=data0[,6]
#查看相关性
corre=cor(data1)
round(corre,3)
观察变量是否适合做主成分分析,约相关约适合

4 R主成分分析 三种方法
4.1方法1 princomp
princomp principal components analysis
data.pca=princomp(data1,scores=T,cor=T)
summary(data.pca)
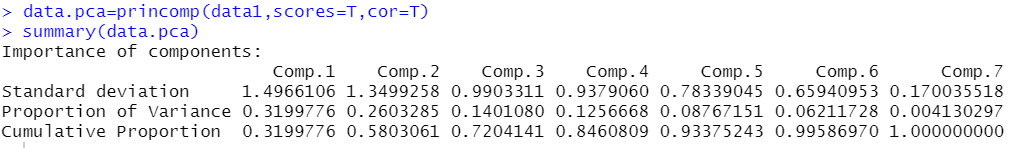
Standard deviation 特征值的开方是 标准差值
Proportion of Variance 方差占的比例
#标准差,实际上是特征值的开方
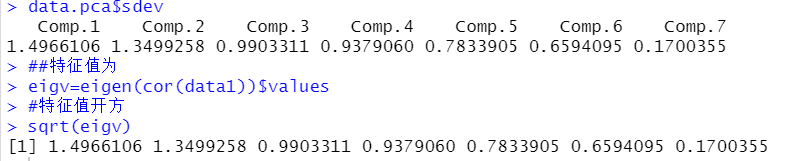
data.pca$sdev
##特征值为
eigv=eigen(cor(data1))$values
#特征值开方
sqrt(eigv)
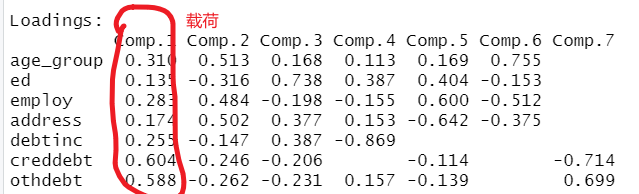
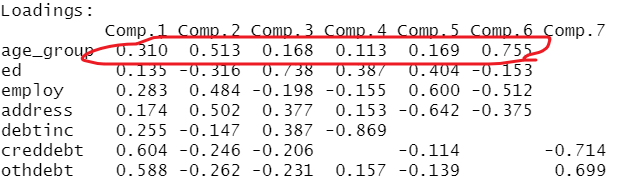
data.pca$loadings #aij 载荷
主成分载荷
因子载荷
#scale ,应该为每个变量的标准差
data.pca$scale
sigmas=apply(data1, 2, sd)
sigmas
主成分打分值,总分是加权和
data.pca$scores
cal_score=scale(data1)%*%data.pca$loadings
cal_score

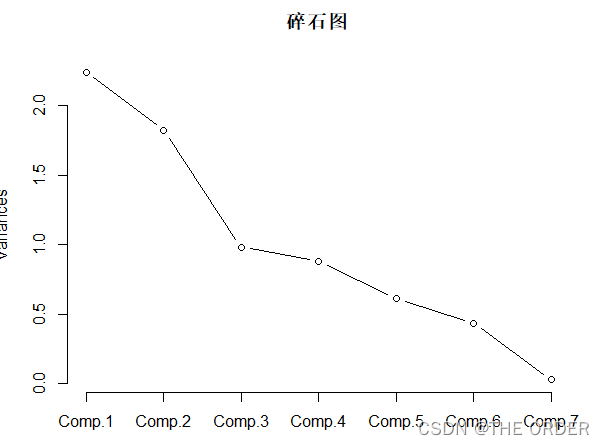
可以通过画图表现主成分的权重筛选
#画碎石图
screeplot(data.pca,type="line",main="碎石图",lty=1)
screeplot(data.pca)
biplot(data.pca)
载荷系数
data.pca$loadings[1,]
data.pca$loadings[,1]
方法2 princomp 未标准化
data.pca.cov=princomp(data1,scores = T,cor=F)
summary(data.pca.cov)
data.pca.cov$loadings
易受到量纲的影响

方法3 用principal 函数对原数据进行分析
pc=principal(data1,nfactors = 7,rotate="none")
pc
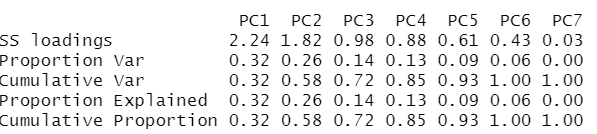
pc$communality
pc$loadings
pc$r.scores#主成分的相关系数
pc$uniquenesses#残差 所有变量,残差接近0
pc$weights
##########################################旋转型
pcr=principal(data1,nfactors = 5,rotate="varimax")
pcr
pcr$loadings#旋转后 系数大的代表相应x的解释信息
round(pcr$scores,3)
主成分的打分结果排序就是y原来的排序
apply(pcr$loadings,2,function(x) t(x)%*%x)
pcr$values
pcr$Vaccounted
pcr$Vaccounted["Proportion Explained",]
score_weights=pcr$Vaccounted["Proportion Explained",]
credit=pcr$scores%*%score_weights
credit
credit1=apply(pcr$scores,1, function(x) weighted.mean(x,score_weights))
credit1
score_sort1=sort(credit1,decreasing = T)
score_sort1
5 主成分检验
#先画碎石图,观察最优主成分个数
ef=fa.parallel(data1,fa="both",n.obs=10,n.iter=100,main="碎石图")
###提取公共因子,未旋转的主轴迭代因子法
efa=fa(data1,nfactors = 2,rotate="none",fm="pa",socores=T,main="碎石图")
efa$loadings
efa$communality
efa$uniquenesses
efa$values
#验证共同度
uni=rep(1,length(efa$communality))-efa$communality
uni
efar=fa(data1,nfactors = 2,rotate="varimax",fm="pa",socores=T,main="碎石图")
efar$communalities
efar$loadings
画图表现主成分
#使用factor.plot()绘制正交或斜交图
factor.plot(efar,labels=rownames(efar$loadings))
#使用fa.diagram()函数之树形图
fa.diagram(efar,simple = F)
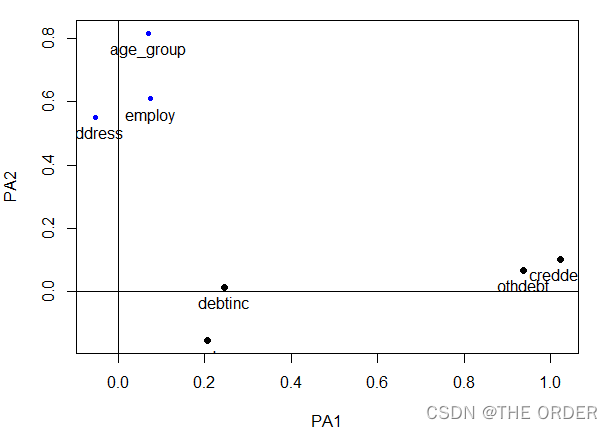
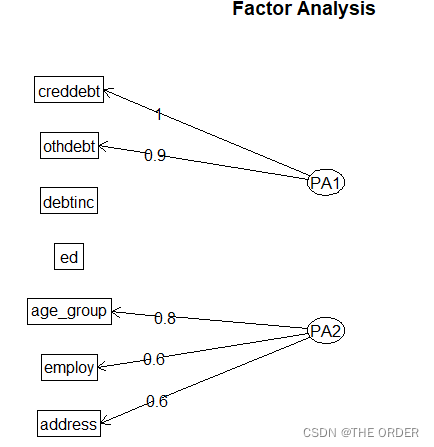
6 其它降维方法
#利用函数进行降维cmdscale {stats}classical
dist(data1)
cMDS=cmdscale(dist(data1),k=2,add=T)
cMDS
【声明】本内容来自华为云开发者社区博主,不代表华为云及华为云开发者社区的观点和立场。转载时必须标注文章的来源(华为云社区)、文章链接、文章作者等基本信息,否则作者和本社区有权追究责任。如果您发现本社区中有涉嫌抄袭的内容,欢迎发送邮件进行举报,并提供相关证据,一经查实,本社区将立刻删除涉嫌侵权内容,举报邮箱:
cloudbbs@huaweicloud.com
- 点赞
- 收藏
- 关注作者


评论(0)