Biopython | 计算蛋白质的接触图(contact map)
【摘要】
contact map
蛋白质接触图使用二元二维矩阵表示三维蛋白质结构的所有可能的氨基酸残基对之间的距离。
计算contact map
导入库
import pandas as pdimport numpy as npfrom Bio import SeqIOimport matplotlib.pyplo...
contact map
蛋白质接触图使用二元二维矩阵表示三维蛋白质结构的所有可能的氨基酸残基对之间的距离。
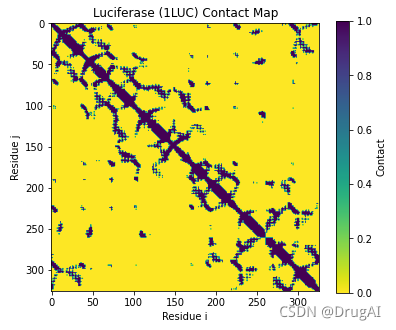
计算contact map
导入库
-
import pandas as pd
-
import numpy as np
-
from Bio import SeqIO
-
import matplotlib.pyplot as plt
-
import requests
-
from sklearn.metrics import pairwise_distances
-
%matplotlib inline
定义坐标提取函数
-
def get_ca_coords(pdb='1LUC', chain='A'):
-
-
url = f'https://files.rcsb.org/download/{pdb}.pdb'
-
r = requests.get(url)
-
r.raise_for_status()
-
-
lines = r.text.splitlines()
-
-
out = []
-
-
for line
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/121118617
【版权声明】本文为华为云社区用户转载文章,如果您发现本社区中有涉嫌抄袭的内容,欢迎发送邮件进行举报,并提供相关证据,一经查实,本社区将立刻删除涉嫌侵权内容,举报邮箱:
cloudbbs@huaweicloud.com
- 点赞
- 收藏
- 关注作者


评论(0)