KDD '22 | 物理模型增强伪标记的 T 细胞受体-肽相互作用预测
今天介绍一篇来由美国NEC实验室Erik Kruus等人于2022年8月在线发表在KDD上的文章。在这篇文章中,作者团队提出通过TCR-肽对的物理建模来扩展训练集,以解决当前数据集中出现的数据稀缺问题。实验证明,用物理建模和数据增强的伪标记来训练深度神经网络,比现有两个数据集中的基线有所改进。

1
介绍
成功预测TCR与肽之间的相互作用是开发个性化药物和疫苗的关键一步,被称为免疫学的圣杯。TCR是具有两条链的二聚体:链和链。每条链具有三个环作为互补决定区(CDR),分别表示为CDR1、CDR2和CDR3。认为CDR3的链有较高的变异,主要负责识别不同的肽段。因此,用于研究TCR-肽相互作用的数据集(VDjdb和McPAS)主要包含TCR的CDR3链序列和肽序列。随着深度学习的最新进展,已经提出了几种用于预测 TCR 肽相互作用的计算方法,然而这些方法主要依赖于可用的标记TCR-肽对,尽管数据库中存在大量公共可用的TCR(没有已知的相关肽)序列。
作者团队提出的方法也是基于深度学习的。然而,他们并没有专注于设计模型的架构,而是强调计算TCR-肽的物理特性(通过利用没有已知相关肽的大型可用TCR数据库)来扩展训练数据集。作者团队提出的方法适用于任何编码TCR和肽序列以进行预测的深度学习方法,该框架也可推广到研究其他蛋白质-蛋白质相互作用。
2
方法
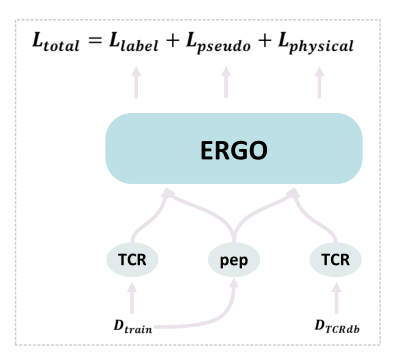
图2:方法概述
作者团队提出的方法训练了一个深度学习模型,用于从3个损失中预测TCR肽相互作用(如图2所示):1)来自给定已知TCR-肽对的监督交叉熵损失;2)基于未知TCR-肽对的对接能量的监督交叉熵损失;3)来自伪标记(通过教师模型)未知TCR-肽对KL-divergence损失。
作者团队使用ERGO-I作为所有实验的基础模型,ERGO-II通过进一步考虑辅助信息,即CDR3的链、V和J基因、MHC类型和T细胞类型。作者选择ERGO-I而不是ERGO-II的原因如下:作者们的目标是验证预测两个分子的相互作用的机器学习模型可以进一步物理建模来改进。
从已知对中学习
ERGO-I有两个独立的编码器:和。TCR的编码器是堆叠的 MLP,并通过自动编码损失进行预训练,而肽的编码器由LSTM参数化(ERGO-II TCR和肽的编码器都是LSTM)。
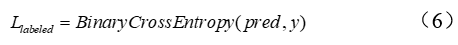
从物理建模中学习
由于训练集中缺乏不同的TCR和肽,作者团队建议利用现有的大量没有相关肽的 TCR 序列,通过对这些TCR和肽之间的物理特性进行建模来扩展训练集。考虑到其简单性,作者团队使用TCR和肽之间的对接能量作为相互作用的指示,以便将其用于大规模未标记的TCR。对接是一种基于物理的建模,首先需要已知的TCR和肽结构。作者团队使用HDOCK对接TCR和肽,通过这种方式,构建了具有对接能量分数的80K TCR-肽对(如图4所示)。然后,作者们将这些能量分数最低的 25% 的对伪标记为正对,将能量分数最高的 25% 的对标记为负对。
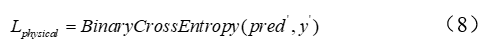
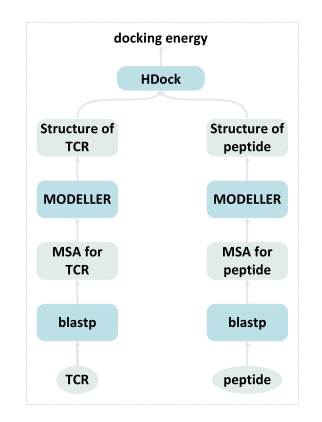
图4:使用 HDOCK 进行对接的概述
从Pseudo-labeled Pairs 学习
除了上一个部分描述的物理建模的伪标签外,还可以利用成熟的半监督方法来进一步改进结果。作者团队首先使用在标记数据集上训练的模型标记未标记示例。然后使用标记的训练集和扩展的伪标记示例重新训练模型(如图5所示)。
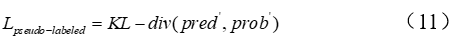
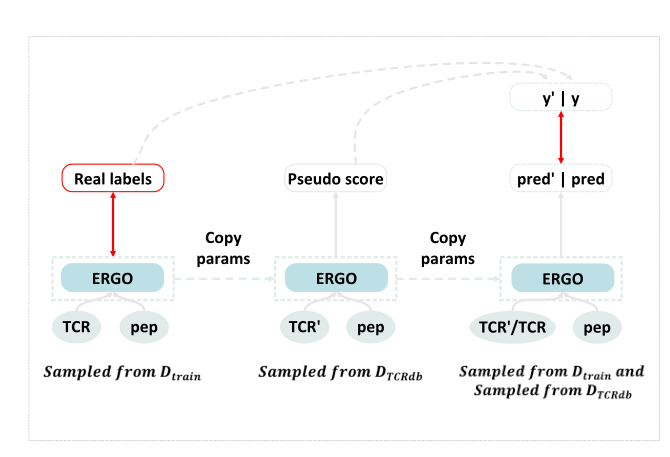
图5:从数据增强伪标签中学习的概述
展望元更新
虽然从物理建模中学习有效地扩展了训练数据集,但学习的成功还取决于物理建模的质量。作者团队希望学习模型,以便针对主要学习目标(测试集上的损失)优化物理建模的辅助学习。这通常是通过最小化验证损失的元学习来完成的。元学习算法引入了一种耗时的梯度梯度学习过程。因此,作者团队借用元学习的想法,而不是最小化验证损失,通过最小化当前批次的训练损失来近似它,即,通过物理建模优化学习梯度,使得来自该辅助目标的梯度仅减少当前批次的训练损失。

3
实验
数据集
作者团队在两个数据集上评估了他们的方法(McPAS和VDJdb)。McPAS是一个手动管理的数据集,包含超过20,000个TCR,匹配超过300个肽段。同样,VDJdb数据集有超过40,000个TCR与大约 200个肽段配对。
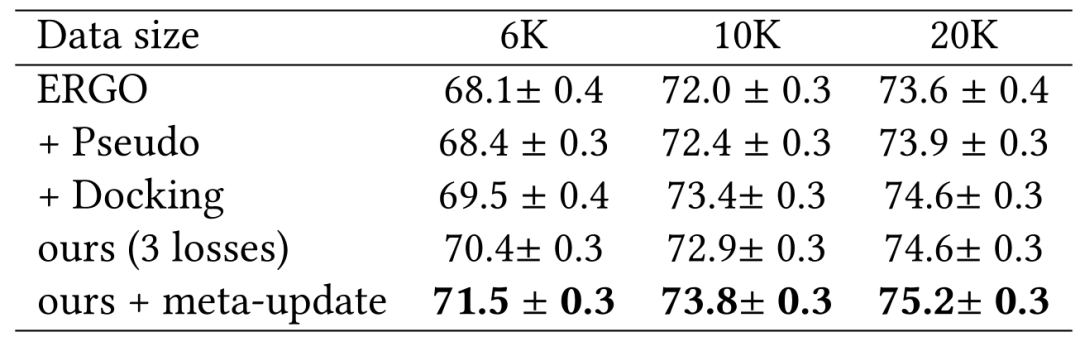
McPAS 上的结果
作者团队在表1和表2中展示了使用ERGO的2种不同变体研究McPAS。即一种使用AE编码器用于TCR,一种使用LSTM用于TCR,对于肽段使用相同的LSTM编码器。表中改进的性能说明了在用数据增强的伪标记和物理建模进行训练期间具有更多样的TCR-肽对的重要性。作者团队还展示了使用双LSTM作为表2中的ERGO 基础模型来改进基线。在这种情况下,数据增强伪标签的效果与物理建模一样好。
表1:基于ERGO-AE模型的McPAS实验结果
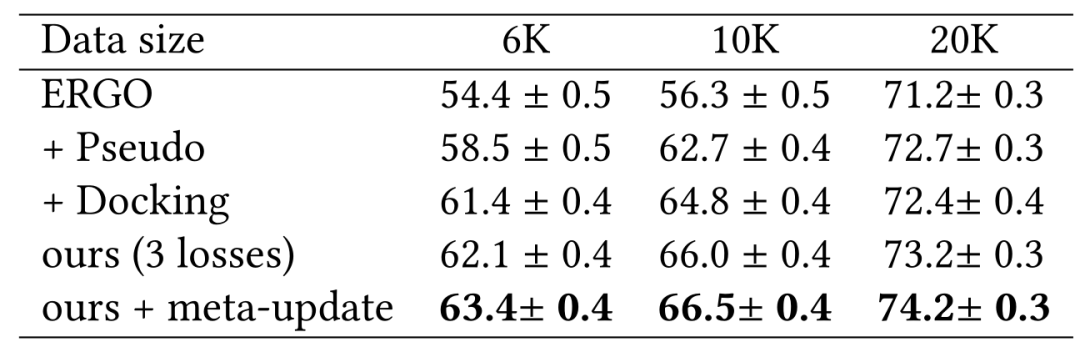
表2:基于LSTM模型的McPAS实验结果
VDJdb 上的结果
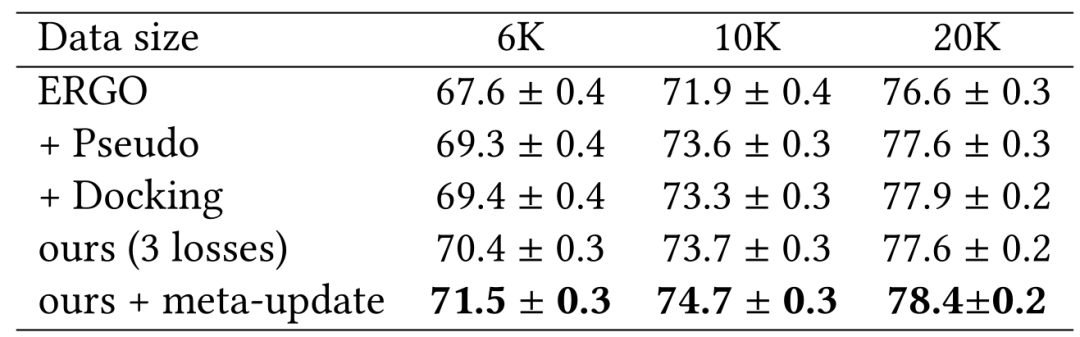
在表4中,作者团队发现数据增强伪标签仅在3个任务中略微优于基线。这可能是由于双LSTM模型的教师模型无法为扩展TCR生成有用的伪标签以重新训练模型。然而,在所有3项任务中,通过对接进行的物理建模始终比基线有了相当大的提高。
表4:使用ERGO-LSTM基础模型在VDJdb上的实验结果
4
结论
在这项工作中,作者团队研究了几种技术来改善TCR-肽相互作用预测。具体地,作者们发现从辅助数据集对未知的TCR-肽对进行伪标记,并用原始数据集和扩展的伪标记数据集的混合物重新训练模型可以改善结果。此外,作为TCR-肽对之间的物理属性的对接能量可以用作训练深度学习模型的替代伪标签。并且通过物理建模的伪标签通常优于通过从原始训练集训练的教师模型的伪标签。
参考资料
https://dl.acm.org/doi/10.1145/3534678.3539075
源码
https://github.com/yiren-jian/Tcell-Peptide-PhyAugmentation
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/126515902
- 点赞
- 收藏
- 关注作者

评论(0)