RDKit:基于RECAP生成片段
【摘要】 尝试使用RECAP进行分子碎裂!
RDKit代码:
#导入模块,载入分子#!usr/bin/python3from rdkit import Chemfrom rdkit.Chem import Recapfrom rdkit.Chem import Drawfrom rdkit.Chem.Draw import IPythonConsolemol = C...
尝试使用RECAP进行分子碎裂!
RDKit代码:
-
#导入模块,载入分子
-
#!usr/bin/python3
-
from rdkit import Chem
-
from rdkit.Chem import Recap
-
from rdkit.Chem import Draw
-
from rdkit.Chem.Draw import IPythonConsole
-
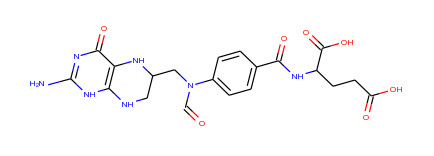
mol = Chem.MolFromSmiles('O=c1nc([nH]c2NCC(Nc12)CN(c1ccc(cc1)C(=O)NC(CCC(=O)O)C(=O)O)C=O)N')
-
#输出分子
-
mol
-
#结果以树结构形式获得
-
node = Recap.RecapDecompose(mol)
-
print (node)
-
#输出叶节点片段
-
leaves = [leaf.mol for leaf in node.GetLeaves().values()]
-
img = Draw.MolsToGridImage(leaves)
-
img.save('leaves.png')
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/81570965
【版权声明】本文为华为云社区用户转载文章,如果您发现本社区中有涉嫌抄袭的内容,欢迎发送邮件进行举报,并提供相关证据,一经查实,本社区将立刻删除涉嫌侵权内容,举报邮箱:
cloudbbs@huaweicloud.com
- 点赞
- 收藏
- 关注作者


评论(0)