RDKit | 基于keras的化合物SeqToSeq Autoencoder
【摘要】 简介
输入化合物结构smiles并创建一个自动编码器模型,该模型将在尺寸压缩后恢复化合物结构smiles。尽管它不是VAE,不能用于生成化合物,但是可以将编码层(图中的压缩表示)用作分子指纹。
数据集
使用最大无偏验证(MUV)数据集(https://pubs.acs.org/doi/10.10...
简介
输入化合物结构smiles并创建一个自动编码器模型,该模型将在尺寸压缩后恢复化合物结构smiles。尽管它不是VAE,不能用于生成化合物,但是可以将编码层(图中的压缩表示)用作分子指纹。
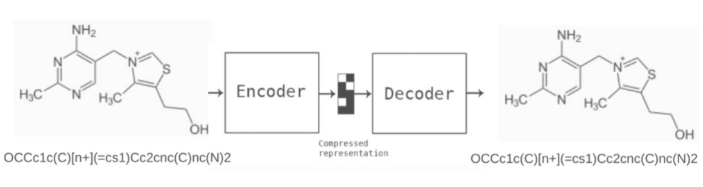
数据集
使用最大无偏验证(MUV)数据集(https://pubs.acs.org/doi/10.1021/ci8002649)。MUV是虚拟筛选基准的约90,000分子,其被设计用于低分子量化合物的数据。
https://github.com/deepchem/deepchem/tree/master/datasets
基于keras的化合物SeqToSeq Autoencoder
数据预处理
导入包
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/103880345
【版权声明】本文为华为云社区用户转载文章,如果您发现本社区中有涉嫌抄袭的内容,欢迎发送邮件进行举报,并提供相关证据,一经查实,本社区将立刻删除涉嫌侵权内容,举报邮箱:
cloudbbs@huaweicloud.com
- 点赞
- 收藏
- 关注作者


评论(0)