Machine Learning | (4) Scikit-learn的分类器算法-逻辑回归
Machine Learning | (1) Scikit-learn与特征工程
Machine Learning | (2) sklearn数据集与机器学习组成
Machine Learning | (3) Scikit-learn的分类器算法-k-近邻
Machine Learning | (4) Scikit-learn的分类器算法-逻辑回归
逻辑回归(Logistic Regression),简称LR。它的特点是能够是我们的特征输入集合转化为0和1这两类的概率。一般来说,回归不用在分类问题上,因为回归是连续型模型,而且受噪声影响比较大。如果非要应用进入,可以使用逻辑回归。了解过线性回归之后再来看逻辑回归可以更好的理解。
优点:计算代价不高,易于理解和实现
缺点:容易欠拟合,分类精度不高
适用数据:数值型和标称型
逻辑回归
对于回归问题后面会介绍,Logistic回归本质上是线性回归,只是在特征到结果的映射中加入了一层函数映射,即先把特征线性求和,然后使用函数g(z)将最为假设函数来预测。g(z)可以将连续值映射到0和1上。Logistic回归用来分类0/1问题,也就是预测结果属于0或者1的二值分类问题
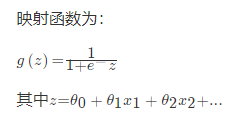
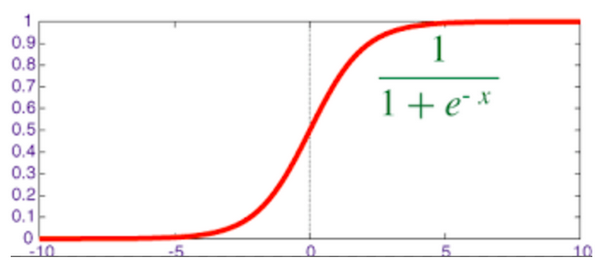
映射出来的效果如下如:
sklearn.linear_model.LogisticRegression
逻辑回归类
-
class sklearn.linear_model.LogisticRegression(penalty='l2', dual=False, tol=0.0001, C=1.0, fit_intercept=True, intercept_scaling=1, class_weight=None, random_state=None, solver='liblinear', max_iter=100, multi_class='ovr', verbose=0, warm_start=False, n_jobs=1)
-
"""
-
:param C: float,默认值:1.0
-
-
:param penalty: 特征选择的方式
-
-
:param tol: 公差停止标准
-
"""
-

-
from sklearn.model_selection import train_test_split
-
from sklearn.datasets import load_digits
-
from sklearn.linear_model import LogisticRegression
-
LR = LogisticRegression(C=1.0, penalty='l1', tol=0.01)
-
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=42)
-
LR.fit(X_train,y_train)
-
LR.predict(X_test)
-
LR.score(X_test,y_test)
-
0.96464646464646464
-
# c=100.0
-
0.96801346801346799
属性
coef_
决策功能的特征系数
Cs_
数组C,即用于交叉验证的正则化参数值的倒数
特点分析
线性分类器可以说是最为基本和常用的机器学习模型。尽管其受限于数据特征与分类目标之间的线性假设,我们仍然可以在科学研究与工程实践中把线性分类器的表现性能作为基准。
逻辑回归算法案例分析
良/恶性乳腺癌肿瘤预测
原始数据的下载地址为:https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/
数据预处理
-
import pandas as pd
-
import numpy as np
-
-
# 根据官方数据构建类别
-
column_names = ['Sample code number','Clump Thickness','Uniformity of Cell Size','Uniformity of Cell Shape','Marginal Adhesion','Single Epithelial Cell Size','Bare Nuclei','Bland Chromatin','Normal Nucleoli','Mitoses','Class'],
-
-
data = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/',names = column_names)
-
-
# 将?替换成标准缺失值表示
-
data = data.replace(to_replace='?',value = np.nan)
-
-
# 丢弃带有缺失值的数据(只要一个维度有缺失)
-
data = data.dropna(how='any')
-
-
data.shape
处理的缺失值后的样本共有683条,特征包括细胞厚度、细胞大小、形状等九个维度
准备训练测试数据
-
from sklearn.cross_validation import train_test_split
-
-
X_train,X_test,y_train,y_test = train_test_split(data[column_names[1:10]],data[column_names[10]],test_size=0.25,random_state=42)
-
-
# 查看训练和测试样本的数量和类别分布
-
y_train.value_counts()
-
-
y_test.value_counts()
使用逻辑回归进行良/恶性肿瘤预测任务
-
from sklearn.preprocessing import StandardScaler
-
from sklearn.linear_model import LogisticRegression
-
-
-
# 标准化数据,保证每个维度的特征数据方差为1,均值为0。使得预测结果不会被某些维度过大的特征值而主导
-
ss = StandardScaler()
-
-
X_train = ss.fit_transform(X_train)
-
X_test = ss.transform(X_test)
-
-
# 初始化 LogisticRegression
-
-
lr = LogisticRegression(C=1.0, penalty='l1', tol=0.01)
-
-
# 跳用LogisticRegression中的fit函数/模块来训练模型参数
-
lr.fit(X_train,y_train)
-
-
lr_y_predict = lr.predict(X_test)
性能分析
-
from sklearn.metrics import classification_report
-
-
# 利用逻辑斯蒂回归自带的评分函数score获得模型在测试集上的准确定结果
-
print '精确率为:',lr.score(X_test,y_test)
-
-
print classification_report(y_test,lr_y_predict,target_names = ['Benign','Maligant'])
参考资料
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/104300688
- 点赞
- 收藏
- 关注作者


评论(0)