Nat.Commun. | DeepAccNet:基于深度学习的准确性估计改善蛋白质结构优化
今天给大家介绍的是华盛顿大学蛋白质设计研究所所长,著名的蛋白质设计天才科学家David Baker课题组发表在Nature Communications上的一项工作。在这项工作中,作者提出了一个深度学习框架DeepAccNet,用于估计蛋白质模型中每个残基的准确性和残基-残基距离中的符号错误,并使用这些预测来指导Rosetta蛋白质结构优化。在Rosetta改进方案的多个阶段中,加入准确性预测,可以大大提高所得蛋白质结构模型的准确性,说明深度学习可以改善对生物分子整体能量最小值的搜索。
介绍
基于氨基酸共同进化数据的距离预测可以显著促进蛋白质结构预测,但是在大多数情况下,预测结构仍然与实际结构有很大差异。蛋白质结构改进的挑战是提高此类起始模型的准确性,迄今为止,最成功的方法是基于物理的方法,该方法涉及大规模搜索低能结构。同时,也有很多基于深度学习和非深度学习的方法,但它们着重于预测每个残基的准确性,而无法预测应如何移动,因此效果都不明显或用处不大。在这项工作中,作者开发了一个基于深度学习的框架——DeepAccNet,该框架可估计每个残基-残基距离中的符号错误以及局部残基接触误差,并使用此估计值来指导基于Rosetta的蛋白质结构优化。
方法
DeepAccNet结构如图1所示,可在蛋白质结构模型的基础上进行三种类型的预测:每个残基的Cβ局部距离差异测试(Cβ1-DDT)分数,阈值为15Å的局部Cβ接触图,以及来自相应自然结构的有符号Cβ–Cβ距离误差的每个残基对分布。作者指出,DeepAccNet不是预测每对位置的单个误差值,而是预测误差的直方图,该直方图提供有关可能结构分布的更详细信息,并更好地表示误差预测所固有的不确定性。
DeepAccNet整合了1D,2D和3D特征,首先在以每个残基为中心的坐标框中对局部原子网格执行一系列3D卷积操作,这些卷积生成描述蛋白质中N个残基中每个残基的局部3D环境特征。这些3D特征以及附加的残基水平一维输入特征通过平铺与2D残基-残基输入特征结合在一起,然后使用ResNet架构将生成的组合2D特征描述输入到一系列2D卷积层中。
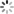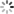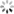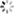
图1 Approach overview
结果
图2显示了对于三个靶蛋白(3lhnA,4gmqA和3hixA),在两个随机选择的诱饵结构上没有MSA或Bert嵌入的DeepAccNet预测。在每种情况下,网络都会为两个诱饵生成不同的有符号残基-残基距离误差图,这些图在质量上类似于结构误差的实际模式。网络还可以准确预测不同诱饵的每个残基模型准确性(Cβ1-DDT分数)的变化, 总体而言,详细的预测提供了有关结构的哪些部分需要移动以及以何种方式指导优化的大量信息。
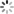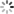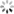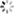
图2 examples of DeepAccNet prediction
为了研究每种特征对网络性能的贡献,作者在训练过程中一次将每种功能与距离图结合在一起,并通过交叉熵损失和测试集上的Cβ1-DDT得分均方误差来评估性能(图3a)。除了MSA特征外,最大的贡献来自基于3D卷积特征和Bert嵌入。作者调查了网络在X射线晶体学,核磁共振波谱(NMR)和电子显微镜(EM)所确定的实验结构上的性能,对于高分辨率晶体结构,DeepAccNet变体的预测Cβl-DDT值接近1.0,而对于较低分辨率的结构,Cβ1-DDT值则有所降低(图3c), 核磁共振结构的预测准确度低于高分辨率晶体结构(图3d)。作者将DeepAccNet变体与其他精度估算器进行了比较(图3b),从最近的CASP实验中可以明显看出,从多个序列比对中得到的协同进化信息提供了详细的结构信息。DeepAccNet-Bert包含Bert嵌入,这些嵌入是由单个序列生成的,没有任何进化比对,在没有同源序列信息的蛋白质上,它在EMA任务上的表现优于DeepAccNet-MSA,当有多个序列比对信息时,DeepAccNet-MSA将是一个更可靠的选择,显示了最先进的性能。
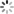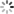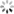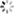
图3 DeepAccNet performance.
作者发现,基于网络的准确性预测会不断提高基准示例的准确性。通过更新的方案,无论蛋白质大小如何,在起始模型上均观察到一致的改善(图4a), 对每个残基模型质量的更详细预测也与实际值非常吻合(图4e)。作者还评估了准确度预测对改进模型质量的实际影响(图4c), 从DeepAccNet 柱状图预测得出的残基对约束对于成功提炼至关重要(图4d)。如图4f所示,改进包括在整体结构正确时识别和修改错误区域,精度预测网络通过两种方式促进了这一总体改进:它提供了对不可靠的距离对和区域的更准确的估计;其次,它提供了一种有效地估计距离的方法。
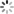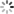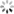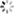
图4 Guiding search in protein structure refinement using the accuracy predictors.
总结
这项工作中,作者提出了DeepAccNet,DeepAccNet对以残基为中心的原子坐标进行3D卷积,将详细的残基信息与其他单独的残基以及残基-残基水平的信息集成在一起。实验结果表明,DeepAccNet可以提供最先进的蛋白质模型精度预测,并且可以将它们进一步用于预测带符号的距离误差,以进行蛋白质结构优化。
参考资料
Hiranuma, N., Park, H., Baek, M. et al. Improved protein structure refinement guided by deep learning based accuracy estimation. Nat Commun 12, 1340 (2021).
https://doi.org/10.1038/s41467-021-21511-x
代码
https://github.com/hiranumn/DeepAccNet.
文章来源: drugai.blog.csdn.net,作者:DrugAI,版权归原作者所有,如需转载,请联系作者。
原文链接:drugai.blog.csdn.net/article/details/114683987
- 点赞
- 收藏
- 关注作者

评论(0)